-
电子病历(Electronic Medical Record,EMR)是指医务人员在医疗活动过程中,使用信息系统生成的数字化信息,包括门(急)诊病历和住院病历[1].电子病历信息抽取可获得与患者密切相关的大量且准确的医疗知识,这些医疗知识在医学问答和辅助决策等方面起着重要的作用[2].
早期的信息抽取将实体识别和关系抽取当作2个独立串联的子任务,忽视了相关性,关系抽取结果的好坏严重依赖于实体抽取的结果,容易产生错误累积的问题,也无法很好地解决实体间关系重叠的问题.近年来抽取工作多考虑将实体识别与关系抽取任务进行联合建模[3-5],构造实体关系联合抽取模型. Bekoulis等[6]将实体识别和关系提取联合起来看作一个多头选择问题,巧妙地解决了实体间关系出现重叠的情况. Li等[7]考虑到实体间的依赖关系将联合抽取任务当作多轮问答处理,很自然地融合了实体抽取和关系抽取2个子任务.
本研究对肾病专科电子病历借鉴Li等[7]的思路完成信息联合抽取,并使用图数据库Neo4j构建肾病专科医学知识图谱,增强医疗服务的便捷性,以期对慢性肾病风险预测研究工作提供数据.
HTML
-
本文对南京市卫生信息中心提供的肾脏病专科874 718条电子病历进行清洗,之后选择120 000条数据进行人工标注,作为实体识别模型和关系抽取模型的训练数据.
电子病历作为医务人员描述患者医疗活动的记录,包含了大量专业术语和习惯用语,如英文检查缩写“Hb”、带单位的检查结果“127.8 ng/mL”以及用正、负号表示的检查项目等.另外,文本的非中文符号因系统设置原因,会出现全角/半角的差异,所以将这些非中文符号统一为半角符号,将英文字符统一为小写,这样避免了同一个字因不同字符产生不同的词向量.
根据肾病相关的医学专用名词将清洗过的文本中的实体分为“药名” “疾病” “症状”等18个类别,再给出所述实体类别之间存在的医学关系,即为实体间关系,共有“患病时长” “治疗方案” “程度”等7种实体关系.
-
本文将实体识别问题转化为整体的序列标注问题,即对一句话中多个词同时进行标记,最终选择标签转移概率最大的标注序列作为结果,相比于传统的机器学习分类方法,该方法有较强的扩展性和适应性.标注方式采用BIO标注法,B表示实体的头部,I表示实体的中间,O表示非实体部分,标注的结果作为实体识别模型的标签.
本文为了避免实体与关系独立抽取产生信息的冗余和联合抽取导致实体识别准确率下降的问题,提出了新的抽取规则:从已标注的序列标签中抽取实体,同时从文本中抽取实体及实体关系,将两者结合得到最终抽取结果,具体步骤如下:
Step1 实体抽取
根据实体边界和实体类别标签抽取出实体,记录实体词首和词尾在文本中的位置,将其定义为实体位置.
Step2 关系抽取
本文将关系抽取看作抽取一个三元组(主实体,实体关系,客实体).
1) 预测出主实体词首和词尾在文本中的位置;
2) 根据主实体的位置序列进一步预测出客实体的位置,同时预测出实体关系类别.
Step3 结合实体抽取和关系抽取
对比step1获得的实体位置和step2得到的三元组和各实体位置,在一个三元组中,若主实体位置与客实体位置同时存在于第一步获得的实体位置中,则保留该三元组,否则删除.
-
在中文文本中单个字的语义非常有限,仅以字向量为输入特征难以储存语义信息.为了增加输入特征的信息,本文实体抽取模型的输入特征由词向量和字向量两部分构成.
词向量通过预训练好的Word2Vec模型构成,Word2Vec采用分布式的低维度、稠密词向量表示,可以充分考虑词的上、下文信息,将语义相似的词映射到向量空间的相近位置,从而保留词汇间的语义信息.为获得更具针对性的词向量,本文使用去除训练模型数据后剩余的肾病电子病历作为词向量训练数据,采用jieba分词工具,并把肾病相关的常用医学专用名词和单位加入jieba自定义词典中对文本进行分词,预训练出一个Word2Vec模型.
字向量由文本以字为单位输入一个随机初始化的字Embedding层得到,字Embedding层实际是个不带偏置的全连接层,以每个字的独热编码为输入,全连接层节点个数即字向量的维数,而这个全连接层的参数,就是一个“字向量表”,它随着模型训练不断优化.在训练模型的过程中只优化字向量,而不改变词向量,保留了词向量的语义信息.
有效的字向量和词向量的结合方式超越了单独的字向量和词向量[8],本文通过加和的方式将词向量与字向量结合,得到实体抽取模型输入特征.从关系抽取方法和对电子病历的分析过程中发现,实体在文本的位置信息具有一定价值,主实体和客实体的相对位置往往不会太远,因此本文关系抽取模型将实体位置特征加入到词向量和字向量中.
-
实体抽取模型分为3层,第1层是词嵌入层,即将文本转化为模型的输入特征,该层获得的字、词加和向量作为第2层的输入;第2层采用BiLSTM模块,其作用是自动提取文本特征.选用2个方向相反的LSTM构成BiLSTM模块可以分别从前向和后向学习词的上文特征和下文特征,将二者拼接得到上下文特征,再将包含上下文信息的文本特征作为CRF线性层的输入;第3层即CRF模块,由于词的标签除了受上下文信息的影响外,还需遵循标注模式的规律,而BiLSTM模型忽略了标签间的依赖关系,加入CRF层可以优化序列标签,从整体出发学习整个文本的标签转移概率,得到遵循标注规律的最优结果.
-
本文关系抽取模型使用高效率的CNN+BiLSTM模型构成联合抽取模型[9].利用CNN抽取词语部件特征中的关键语义特征,丰富字词级别的语义信息[10]. BiLSTM获得上下文特征,通过抽取到的特征预测出主实体,再由主实体同时预测出客实体及实体关系,这样的抽取方式有效地解决了关系重叠的问题.模型实际输出结果是一个长度与输入相同的0/1序列,预测主实体的输出结果是在主实体词首和词尾处标注为1,其余地方标注为0,预测客实体及关系的输出结果是在客实体词首和词尾处标注实体关系类别的编号,其余地方标注为0.
CNN的架构由3个不同的层组成:卷积层、最大池化层和全连接层.本研究中实体词数基本大于2个字符,文本平均长度大于300个字符.为了尽可能多地提取上下文特征,扩大感受野,模型参考了Wang[11]等的报道,把CNN中的普通卷积换成了空洞卷积,空洞卷积使感受野得到指数级扩展,同时不会损失分辨率或覆盖范围[12-13].最大池化层可以在保持词序列属性的同时减少神经网络的参数,从而有效防止模型过拟合[14].
1.1. 数据预处理
1.2. 实体与关系的抽取
1.3. 构造输入特征
1.4. 实体抽取模型
1.5. 关系抽取模型
-
构建慢性肾脏病专业数据库,库中存储慢性肾脏病专业书籍与文献,再根据慢性肾脏病专业数据库人工构建一个包含血液检查项目、尿液检查项目、症状及其他医学实体名词的标准库,标准库中涵盖每个医学名词的标准名称及出现过的相似名称,并对每个医学名词进行编码便于唯一识别,形成实体标准库.
-
模型抽取得到的实体中,同一种实体的不同表述对照2.1节构建得到的实体标准库进行替换,将符号、字母、文字、单位、医学代码统一采用实体描述,得到标准化的实体数据.
-
在目前的医学领域中,对基于自然语言处理的医学知识图谱进行研究,常用Neo4j图数据库构建医学知识图谱,如构建临床合理用药知识图谱库[15]以及在中医药方面对知识图谱的可视化分析以及检索的研究[16]等. Neo4j具有高性能、设计灵活、开发敏捷性等优势[17],Neo4j图数据库将结构化数据存储在网络上,能够更便捷地展示不同实体之间的关系.
本文将模型抽取得到的实体类型、实体关系和标准化后的实体数据以三元组的形式输入Neo4j图形数据库平台,构成肾病专科知识图谱.
2.1. 构建数据库
2.2. 实体标准化
2.3. Neo4j知识存储模型
-
本文对南京市卫生信息中心提供的肾脏病专科874 718条电子病历中的120 000条数据进行人工标注,再将文本按句拆分成166 148个句子,作为实体识别模型和关系抽取模型的建模数据.将166 148条数据随机打乱,按8:2的比例分为132 918条训练数据和33 230条测试数据.
-
使用BiLSTM+CRF模型做实体识别与类别抽取,设置输入向量维数为128维,优化算法使用自适应时刻估计方法(adaptive moment estimation,Adam),损失函数选用交叉熵损失函数,学习率设置为0.001,dropout设置为0.25,模型预测出文本的BIO序列,将识别出的实体及类别与实际实体及类别对比,计算精确率和召回率,进一步计算F1值,最终以F1值作为模型评估标准.其中,表示实体识别模型对17种实体类型精确率和召回率的评估如表 1所示.
表 1结果显示各实体类别的精确率都不低,但是召回率结果参差不齐,过敏史、体质量、手术、身高、BMI的召回率结果都低于50%,但是精确率都非常高,分析原因可能是因为这些实体类别在肾病电子病历中出现频率过低,支持样本太少且在样本中的结构和语法相近,导致泛化能力弱,文本结构稍有变化模型就无法识别.对于疾病和症状可能是因为在电子病历中的位置很相似导致分类失误.
由于电子病历非常具有针对性,对不同的患者有不同的健康信息,因此不同的文本其实体类别的分布非常不均,例如“过敏史” “手术”等非普遍型实体类别占总数据量不到0.01%,属于支持样本过少的实体类别.
-
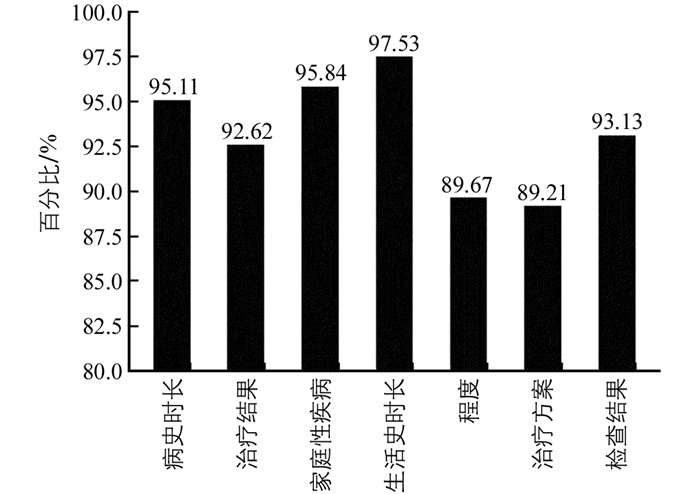
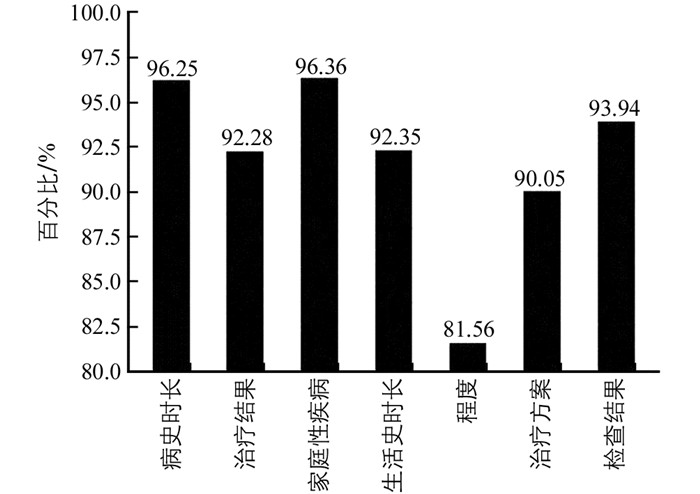
关系抽取采用CNN+BiLSTM模型,设置输入向量维数为128维,最大字符数为512,优化算法使用自适应时刻估计方法(adaptive moment estimation,Adam),选用主实体的二分类交叉熵与客实体及实体类别的多分类交叉熵之和作为损失函数. 7种实体关系精确率和召回率结果如图 1、图 2所示.
图 1结果表明各实体关系的精确率都很高,图 2结果表明“程度”的召回率低于其他6种实体关系,原因可能是构成此关系的主实体和客实体相对位置多变,且语法较复杂,模型不能很好地学习结构较复杂的文本.
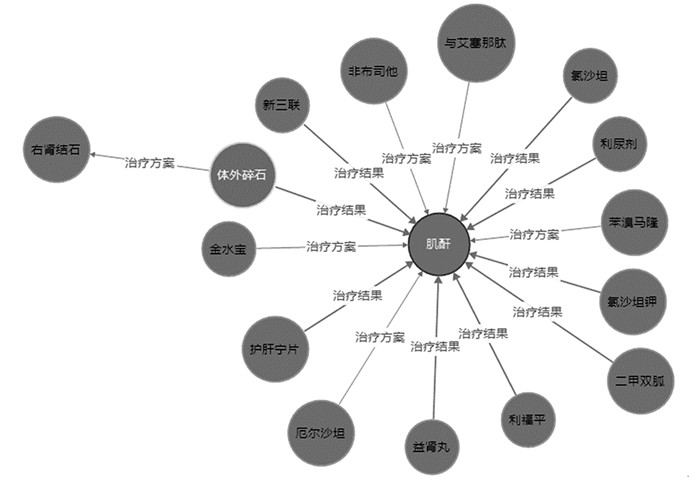
虽然模型取得了不错的效果,但仍存在不足,如多个实体间存在关系重叠时使用的是就近原则,一定程度上降低了结果的准确率.另外,各实体类别支持样本参差不齐,影响了较少样本实体类别的识别,进一步也影响了实体关系的识别.可以尝试使用注意力机制同时扩充数据,提高召回率.本文的知识图谱仍需进一步完善,以便识别新实体类别和关系,最终获得完备的肾病医学知识图谱(图 3).
-
根据具体疾病类型对肾病电子病历进行分类,对不同疾病的肾病电子病历文本抽取得到的实体、实体关系数据格式化,构成(主实体,实体关系,客实体)三元组的形式,传送到Neo4j本地数据库中作为知识图谱三元组,构建一个与具体疾病相关联的肾病医学知识图谱.用户通过输入具体肾脏疾病,可以获得与该疾病相关的检查、症状及治疗手段,能够提醒用户需要注意的检查指标,并根据症状程度、检查结果等辅助判断是否有相关病症,同时也能方便医务人员查阅相关疾病,起到辅助医疗作用.
图 3是Neo4j数据库中构建的知识图谱截取,图的中心为检查实体节点:“肌酐”,导致肌酐异常的药物和治疗肌酐的药物与肌酐构成关系对(如:体外碎石->肌酐关系对表示手术体外碎石可导致肌酐异常,二甲双胍->肌酐关系对表示药物二甲双胍可治疗肌酐异常).疾病节点“右肾结石”与手术节点“体外碎石”构成的关系对表示右肾结石可采用体外碎石的治疗方案.知识图谱将疾病与具体治疗方案、不同方案可能产生的结果以及后续治疗方案的选择做了可视化处理,各实体间的关系一目了然,可为肾病临床治疗提供思路.
3.1. 实体识别结果分析
3.2. 关系抽取结果分析
3.3. 知识图谱结果分析
-
本文采用现有方法来构建肾病专科医学知识图谱展示肾病大数据,使用信息抽取研究领域较成熟的BiLSTM+CRF模型识别文本中的肾病相关医学实体,又用联合抽取的方式抽取实体关系,最终将结果保存到Neo4j数据库中并可视化,构建肾病专科医学知识图谱,清晰地展现了不同肾病对应的症状、检查等重要信息.肾病专科医学知识图谱为循证医学研究和疾病监控等提供支持,同时让患者更直观地了解自己的病情,能针对症状和检查结果通过知识图谱进行初步自诊,也帮助医务人员快速查阅疾病信息,使肾病关系变得明朗,达到辅助肾病诊疗的作用,对发展智慧医疗具有一定的实际参考意义.





 DownLoad:
DownLoad: