-
开放科学(资源服务)标识码(OSID):

-
云南黄连(Coptis teeta Wall.)俗称云连,是产自高黎贡山的重要药用植物,含小檗碱、药根碱和其他化合物[1-2]。云南黄连在我国主要分布在云南的西北部腾冲、福贡、维西、德钦等地[3]。云南黄连繁殖方式兼具有性生殖和无性繁殖,具有高度杂合性[4-5]。自然传播和人为传播使得云南黄连积累了自然演化和人工选择的变异,形成了多样性的云南黄连资源[6]。云南黄连在居群内和居群间均存在丰富的表型变异[7]。
目前,云南黄连的研究主要集中在资源调查、栽培技术、传粉生态学、成分含量、药理等方面[5, 8-12]。伴随着分子标记技术的发展,在黄连属物种的遗传多样性、亲缘关系和物种鉴定方面取得了一定进展。早期随机扩增多态性DNA(Random Amplified Polymorphic DNA,RAPD)、简单重复序列间扩增(Inter Simple Sequence Repeat,ISSR)、相关序列扩增多态性(Sequence-Related Amplified Polymorphism,SRAP)标记被开发用于研究黄连属种间和种内的遗传多样性[13-15]。伴随着测序技术的发展,基于转录组序列开发了黄连属的简单重复序列(Simple Sequence Repeats,SSR)标记[16]。常用的叶绿体和细胞核条形码DNA的基因片段被开发用于鉴定黄连属的种间亲缘关系和进化关系分析[17-18]。然而,这些标记均未能有效鉴别黄连属的种间差异,难以用于品种鉴定,不利于黄连种质资源的保护、开发和利用。DNA分子身份证可用于快速准确地鉴定种内不同的个体,能为种质资源的鉴定和保护提供科学依据。构建DNA分子身份证的分子标记主要有SSR和单核苷酸多态性(Single Nucleotide Polymorphism,SNP)。SNP标记作为第三代分子标记,多态性位点数量庞大,能有效地区分不同种质资源。目前,基于SNP构建指纹图谱或者分子身份证的作物越来越多,包括水稻[19]、玉米[20]、大豆[21]、肉苁蓉[22]、山核桃[23]等。
云南黄连资源存在种群间差异。过去由于不规范引种及频繁交换传播,造成生产上同名异物或同物异名的现象,不仅给云南黄连资源的收集保护利用带来诸多困难,也损害了消费者的权益。云南黄连传统的鉴定依据主要是叶片的形状、小叶的疏密程度及花萼的长度等形态学性状。不同种群间存在特征性状交叉重叠,难以快速准确地鉴别。黄连属的基础研究薄弱,尚未见开发SNP标记和分子身份证构建的研究报道。因此,本研究拟对来自云南高黎贡山不同区域的5个云南黄连群体开展简化基因组重测序,开发SNP标记,构建DNA分子身份证。研究将有助于区分不同的云南黄连种群,为黄连属的物种分类、品种鉴别、遗传演化、遗传多样性等研究提供技术支持和理论依据。
全文HTML
-
供试材料为采自云南高黎贡山区域的5个云南黄连群体,采集信息见表 1。
-
-
武汉基诺赛克科技有限公司负责云南黄连基因组DNA提取及质量检测。使用北京全式金生物技术有限公司开发的植物基因组DNA提取试剂盒(EE111)提取云南黄连叶片基因组DNA。采用1%琼脂糖凝胶电泳和Nanodrop检测基因组DNA的完整性和纯度。使用荧光计Qubit 3.0进行核酸定量,浓度大于50 ng/μL,总量大于2 μg。
-
在武汉基诺赛克科技有限公司对提取的基因组DNA进行简化基因组测序,流程包括文库构建、文库质控和上机测序。
1) 文库构建
群体基因组DNA检测合格后,严格按照武汉基诺赛克科技有限公司开发的ddRADseq流程进行文库构建。将200 ng基因组DNA用限制性内切酶MseⅠ和SacⅠ(New England Biolabs公司)完全酶切。将酶切产物用自主设计的特异性接头连接后取特定量的连接产物进行等量混合并用磁珠(AMPure XP公司)纯化。用高保真聚合酶KOD-Plus-Neo(TOYOBO公司)进行PCR富集。将所有产物等量混合并用低熔点的琼脂糖(Bio-Rad公司)低压过夜电泳,选取300~400 bp的范围,然后用琼脂糖凝胶回收试剂盒(QIAGEN公司)进行纯化。
2) 文库质控
使用荧光计Qubit3.0进行初步定量。使用Agilent 2100对文库的插入片段进行检测,插入片段符合预期且无接头污染后才可进行下一步实验。用QTOWER实时荧光定量PCR仪(ANALYTIKJENA公司)对文库的有效浓度进行准确定量,有效浓度不低于2 nmol/μL为合格文库。
3) 测序及数据质控
按照目标下机数据量对文库进行混合,用Illumina HiSeq平台进行Paired-end 150 bp(PE150)测序。将得到的原始图像数据文件经过碱基识别(Base Calling)分析转化为原始测序序列(Raw Reads),结果以FASTQ(简称为fq)文件格式存储。碱基的质量数以不同的字符来表示(每个字符对应的ASCII值减去33,即为对应碱基的质量数)。如果Illumina HiSeq的碱基质量数用Q表示,测序错误率用E表示,则Q=-log(E)。对测序得到的原始测序序列进行质控过滤以后得到的序列(去掉接头的序列,Clean Reads),使用软件fastp 0.23.0去除原始测序序列中含有的接头序列及低质量碱基(以4 bp的滑动窗口计算平均质量数,若碱基质量数低于15则去除其后的所有碱基);原始测序序列的长度必须大于50 bp。
-
Stacks高通量测序软件根据测序序列的相似性生成测序序列的标签,每个标签相当于一个ddRAD测序位点,然后比较不同群体间的测序序列的标签来开发多态性标记。对Stacks的原始输出结果进行筛选获得可用的SNP分子标记。
-
使用软件MEGA 7中的邻接法构建系统发生树,并使用软件ggtree 1.7.10进行可视化。
-
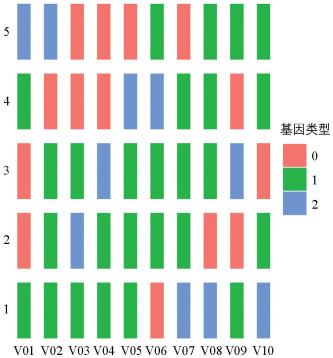
对筛选获得的能够区分所有群体的10个SNP数据进行数字编码,作为构建分子身份证的基本信息。SNP标记有0/0、0/1和1/1共3种基因型,分别用0、1和2表示,10个SNP就可以获得10位数字的编码,以群体1为例,在V01处的基因型为0/1,对应编码为1。以此类推,将基因型全部转换成10位数字编码1111102212,将数字编码转化成二维码。
1.1. 材料
1.2. 方法
1.2.1. 基因组DNA提取及质量检测
1.2.2. 简化基因组测序
1.2.3. 多态性标记的开发
1.2.4. 系统发生树分析
1.2.5. 分子身份证构建
-
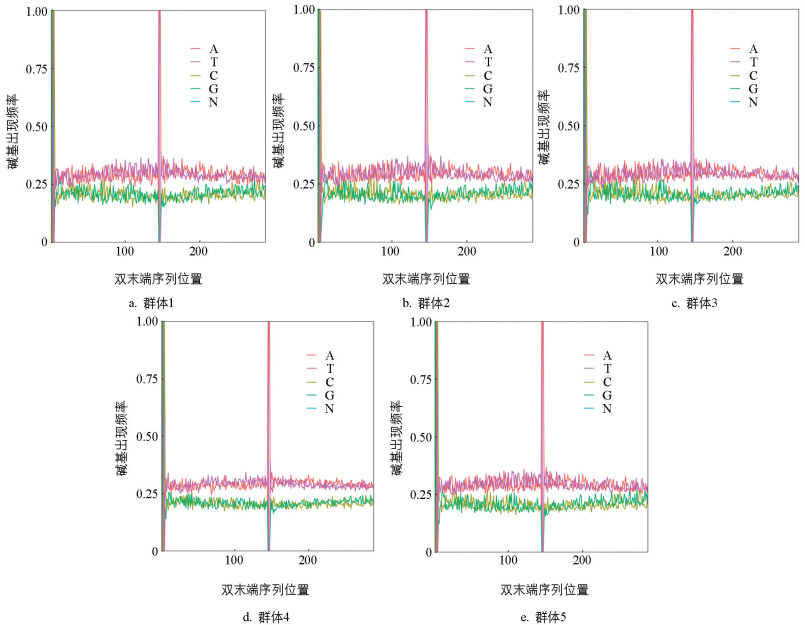
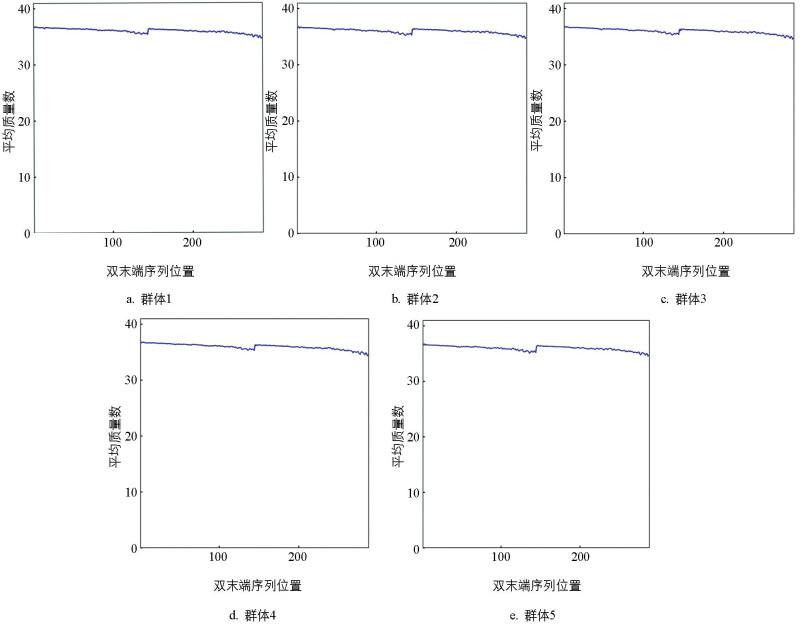
为了保证后续分析的正常进行,测序数据的碱基质量数应该主要分布在30以上。结果表明:5个群体测序数据碱基质量数均分布在30以上(图 1)。测序数据中碱基A、T、C、G、N的分布是评估数据质量的重要指标。N的含量反映了测序质量的好坏。从图 2、表 2看出,5个群体单核苷酸GC含量平均值为41.30%,Q20和Q30平均含量分别为98.12%、94.10%,N含量接近0,因此,测序数据中碱基含量分布及质量分布均符合预期要求。
-
比较不同云南黄连群体间标签来开发多态性标记,统计每个群体中标签的数量以及平均测序深度(表 3)。5个群体平均测序标签数为140 908,平均测序深度为41.42。对Stacks的原始输出结果进行筛选后,共获得34 891个可用SNP分子标记(表 4),表明云南黄连具有丰富的分子变异位点,这为云南黄连的SNP分子标记的开发、遗传多样性分析、遗传演化分析等研究提供了理论参考。
-
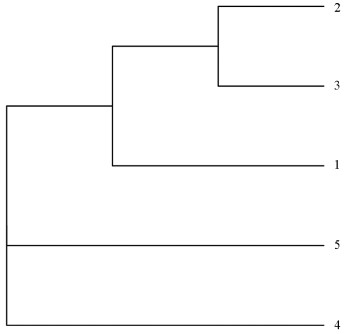
为研究5个云南黄连群体的进化关系,基于测序后获得的所有有效的SNP位点,使用MEGA 7构建系统发生树,并使用ggtree 1.7.10进行可视化(图 3)。结果表明,群体2(福贡县鹿马登乡)和群体3(福贡县石月亮乡)聚在一起,表明这两个群体亲缘关系较近,可能是起源于同一个居群,再传播至不同地方;群体4(福贡县上帕镇)和群体5(泸水市老窝镇)各聚为一类,说明它们的亲缘关系较远,群体1(贡山县独龙江乡)与群体2、3亲缘关系相对较近,与群体4、5较远。
-
在染色体不同位置选择能够区分5个群体的10个SNP标记(表 5),基于其序列信息组合,分别构建了5个群体的分子身份证(图 4),并生成了每个群体的二维码(图 5)。这10个SNP标记具有较好的基因分型效果。群体2和群体3在V01、V02、V05、V06、V07标记中表现出同样的基因分型,与其他群体相比,它们之间有更多的相似性,这与系统发育树的结果一致。开发的10个SNP标记后续可用于云南黄连的种质鉴别、亲缘关系分析、亲本选择等,方便查看各种质的遗传信息。
2.1. 测序数据质控与统计
2.2. 多态性标记的开发
2.3. 系统发生树分析
2.4. DNA分子身份证构建
-
本研究利用高通量的简化基因组测序技术获取了云南黄连大量的多态性SNP变异位点,基于10个SNP标记成功构建了5个云南黄连群体的分子身份证,有助于后续种源鉴定和真伪鉴定。基于序列信息构建的系统发育树也成功地揭示了5个群体的不同亲缘关系,为云南黄连的演化和地理分布特征提供了参考。
过去,由于黄连属的遗传基础研究薄弱,大多采用传统的表型鉴定方法来区分不同物种。但是这种方法不仅需要较强的植物学分类能力,且较难用于区分种以下水平的基因型差异。近年来,基于测序的分子标记技术在黄连属物种鉴别中的应用有所增加。文献[24]采用PCR和测序技术获取黄连属4个种和1个变种(黄连、峨眉黄连、三角叶黄连、云南黄连、短萼黄连)共计230株个体的4个基因片段(rbcL4,ITS,trnH-psbA和matK)的序列信息,但是仅能将云南黄连与其他种区分,ITS的鉴别能力仅为20%~40%。可见,这4个常用的DNA条形码片段信息难以区分黄连属种间差异,同时也说明了云南黄连与其他物种间有较明显的差异。文献[25]采用psbA-trnH、ycf1、ITS和ETS序列信息鉴别来自世界的16个种和3个变种共计39份黄连属群体,其中ITS+ycf1组合具有高达79%的分辨率,开发的条形码成功地鉴别了黄连、云南黄连和三角叶黄连。但是,有个别物种仍无法鉴别,且种以下水平的材料鉴别依然较难。可见,基于少数几个基因片段的测序分析并不能满足黄连属的物种精准鉴别的需求。本研究首次成功地采用SNP分子标记有效地鉴别了黄连属中云南黄连不同地理区域的5个基因型的材料,这对黄连属种以下水平的物种鉴别具有重要的指导作用。此外,基于所有SNPs位点构建的5个云南黄连群体的系统发育树聚类的结果与基于10个SNP基因分型数据构建的分子身份证编码的结果一致,可见筛选的10个SNP标记具有较好的代表性,可作为其他黄连物种的分子分类依据。
2016年我国公布的《植物品种鉴定DNA指纹方法总则》[26]优先推荐SSR和SNP标记用于构建植物指纹图谱[27]。近年来,由于简化基因组测序技术成本的下降,SNP分子标记以高通量、分型稳定等优点逐渐成为农作物品种真实性鉴定、指纹图谱和身份证构建的主流方法[28]。竞争性等位基因特异性PCR(Competitive Allele Specific PCR,KASP)是由英国LGC公司开发的SNP基因分型技术,使用起来更加便捷,成本更低,已发展成为全球标杆技术[29]。目前,基于SNP开发的KASP技术已广泛应用于物种的基因分型、指纹图谱和身份证构建、遗传多样性分析等[27, 30-31],SNP标记转化为KASP标记后使用起来更加便捷,也为本研究的后续开发应用提供了重要思路。本研究成功地开发出10个核心SNP标记,并直接用于构建云南黄连的分子身份证,后续可将SNP标记转化成KASP标记,用于鉴别云南黄连的真伪。
然而,群体数量和地理覆盖范围的限制可能会制约SNP标记的普适性应用。本研究的云南黄连作为中国西南地区高黎贡山的特有药用植物,野生种群受地理隔离、生境破碎化影响,可能导致与其他地域的居群间存在显著遗传分化。5份样本也难以覆盖该地区云南黄连物种水平的等位基因多样性,可能遗漏低频但具有种群特异性的SNP位点,导致分子身份证在区分近缘居群或个体时出现“标记盲区”。筛选出的SNP标记可能只是富集局部适应性变异,而对其他地理单元的鉴别能力较弱。因此,后续将在保护生物学原则的基础上,非损伤性采样收集高黎贡山不同海拔更多地域的云南黄连DNA群体,采用SNP分子标记进行群体遗传结构的分析,深度挖掘该地区云南黄连的核心变异区域。结合其他地区的黄连资源,进一步筛选跨居群通用的核心SNP。
此外,SNP标记不仅可用于构建分子身份证,还可用于挖掘功能基因。在玉米、水稻等大宗作物上定位了大量与重要农艺性状、抗逆性及品质相关的SNP位点[32-36],并挖掘出关键功能基因。云南黄连的药效成分(如小檗碱、巴马汀)合成依赖复杂的代谢通路,将来也可通过高密度SNP标记的全基因组关联分析并快速定位药用成分,合成相关的功能基因并解析其遗传机理,为云南黄连的遗传改良提供分子靶点。
-
利用简化基因组重测序技术获得大量的云南黄连SNP差异位点,基于SNP序列成功构建了5个云南黄连群体的系统发育树,筛选出10个核心SNP标记用于构建云南黄连的分子身份证及二维码,为后续的不同云南黄连资源的鉴别提供了技术支持。



 下载:
下载: