-
随着环境不断的变化,多细胞生物体经历着细胞的增殖、膨胀和分化的基本过程,促使其连续不断地生长和发育[1].尽管发育中的器官最终大小受周围环境条件刺激的影响,但其拥有内在的遗传因子决定了它们最终的大小[2-4].在动物中,器官的大小是由多种多样的细胞生长、增殖和细胞凋亡过程调控的.那些过程主要由雷帕霉素靶蛋白途径和Hippo信号通路调控[5-6].在植物器官形成期间,器官生长由细胞增殖和细胞扩增调控,达到其特征尺寸是由细胞数量或细胞大小决定或两者同时决定的[2-3].
在植物中,主要通过促进细胞增殖而达到促进植物器官生长的调控因子有ANT,JAGGED /JAG,ARGOS,AtGRFs,AtGIFs,KLUH/CYP78A5和GLUCOSE-6-PHOSPHATE TRANSPORTER/GPT2[4, 7-8].而通过限制细胞增殖负调控器官大小的调控因子有CIN,ARF2,PPD1/2,STERILE APETALA/SAP和CINNAMOYL CoA REDUCTASE/CCR1[9-11].同时,植物器官大小也被细胞扩增调控.目前,已经报道有大量通过促进细胞扩增而达到调控器官生长的调控因子,如ROT3,AN,ARL,SAUR,OSR2,KUA1,AtEXPA1,AtEXP10,POM-POM2,ABP1等[12-15].而通过负调控细胞扩张而控制器官生长的几个调控因子有BPEp,RPT2a,OsMPS,AtPRX71,Sl-IAA17,KNOPE1,AtKIN13A和RALF[15-18].
另外,已经报道了几个同时调控细胞扩增和细胞数量的调控因子,这些调控因子被证明可调控器官的大小,如CYCA2/1,DA1,MED25/EOD1/BIG BROTHER,SUPPRESSOR OF DA1/UBP15[19-21].然而,细胞增殖和细胞扩增能够相互补偿,影响最终的器官大小.在这种情况下,植物器官大小是由细胞增殖和细胞扩增协同或反对(向)决定的.现在已鉴定出一些植物响应内在或环境诱导的调控因子,这些调控因子通过协同细胞数量和细胞扩增从而调控器官大小.例如,光敏色素B(Phytochrome B)和光敏色素互作因子(PIFs)[1].
在本课题组的前期工作中,通过图位克隆技术分离了一个水稻器官变小且伴随叶片早衰基因OsROSES1(Reduced Organ size with Early Senescence),利用细胞学观察、生理学测定分析突变体roses1表型特征,并构建由ROSES1启动子驱动的GUS信号表达载体,利用根癌农杆菌(Agrobacterium tumefaciens)介导转化法获得了转基因植株.表型观察结果表明,ROSES1作为水稻BEL1-Like“同源盒基因”通过调控细胞增殖和扩张从而控制器官大小和光合作用,在植物器官发育过程中具有重要调控作用[15].本研究拟通过RNA干涉(RNA interference,RNAi)方法[22]下调OsROSES1基因的表达,以进一步探讨其在水稻生长发育调控中的功能.
全文HTML
-
本研究粳稻品种中花11种子由西南大学农学与生物科技学院作物品质改良重点实验室提供.水稻籼型恢复系涪恢9802种子由重庆市渝东南农业科学院提供.载体pMD19-T购于大连TaKaRa公司.大肠杆菌(Escherichia coli)菌株DH5α,根癌农杆菌(Agrobacterium tumefaciens)EHA105,植物表达载体pCAMBIA1301,pTCK303等由西南大学农学与生物科技学院作物品质改良重点实验室保存.植物RNA提取试剂盒、反转录试剂盒、基因组DNA提取试剂盒、质粒提取试剂盒和琼脂糖胶回收试剂盒购于北京全式金公司. DNA聚合酶、限制性内切酶、T4连接酶、实时PCR试剂盒购于TaKaRa公司.引物设计采用Primer Premier 6.0软件[23],由上海英骏公司合成.序列测定由北京华大基因公司完成.
-
OsROSES1与其他相关同源蛋白的比对、分析和下载在NCBI(http://blast.ncbi.nlm.nih.gov/Blast.cgi),Phytozome 10.3(http://phytozome.jgi.doe.gov/pz/portal.html)和UniProt(http://www.uniprot.org/)网站上进行.进化树的构建使用MEGA 7.0进行[24],各蛋白氨基酸序列的比对在ClustalW上进行,进化树的构建使用邻接法(Neighbor-joining)进行,采用Poisson model,Bootstrap值为1 000次重复.
-
在NCBI网站(https://blast.ncbi.nlm.nih.gov/Blast.cgi)上对OsROSES1基因的编码序列进行BLAST,确定OsROSES1基因的特异区间,在特异区间内设计一对干涉引物用于扩增干涉片段,干涉片段长度为269 bp,位于OsROSES1基因的编码框内,在用于扩增正向片段的引物两端加上KpnI和SpeI酶切位点,在用于扩增反向片段的引物两端加上BamHI和SacI酶切位点,引物5′端均加上GCC保护碱基,引物Ri-ROSES1序列为5′-GGTACCCTCCGCGTGGTCGTACATG-3′和5′-GGATCCCTATGTCCCACTCCCAGGGTTT-3′.以野生型的cDNA为模板,用这两对引物扩增正反向干涉片段,1%琼脂糖凝胶电泳检测并回收,使用相应的内切酶酶切回收产物后-20 ℃保存备用;使用KpnI和SpeI酶切pTCK303载体并回收酶切后的载体骨架,T4连接酶连接正向片段的酶切片段入载体骨架中,转化后挑单斑测序,提取测序正确菌液的质粒作为中间载体,采用BamHI和SacI酶切中间载体,连接反向片段的酶切产物入中间载体转化后挑单斑测序,提取测序正确菌液的质粒即为OsROSES1基因组干涉载体,-20 ℃保存备用.
-
由农杆菌介导OsROSES1基因组干涉载体转化野生型的愈伤组织并经过分化获得OsROSES1基因的干涉植株,转化及分化过程由武汉伯远公司操作完成,转基因植株经过GUS活性检测后种植于西南大学水稻基地,提取野生型、OsROSES1 RNAi转基因T0代植株基因组DNA,利用pTCK303载体上的潮霉素抗性标记Hpt基因序列(Genebank登录号:E00777)进行PCR检测.用扩增引物HPTF:5′-TCGTTATGTTTATCGGCACTTTG-3′和HPTR:5′-GCGTCTGCTGCTCCATACAAG-3′鉴定转基因阳性植株.
-
为了检测OsROSES1基因干涉对植株田间性状的影响,在分蘖期测定野生型、3个T1代RNAi转基因株系的株高、叶片卷曲度、叶片光合色素质量分数,并观察叶片气孔密度和开度.在成熟期测定转基因植株株高、有效穗数、结实率和千粒质量.
-
分别从野生型涪恢9802和roses1突变体抽穗期植株根、茎、节、叶和颖花中提取总RNA,合成第1链cDNA,利用实时定量PCR进行OsROSES1基因组织差异性表达分析.以ACTIN(GeneBank登录号为X16280)作为内参基因,所用实时定量PCR引物为RTACTF:5′-GACCCAGATCATGTTTGAGACCT-3′;RTACTR:5′-CAGTGTGGCTGACACCATCAC-3′;Ri-ROSES1F:5′-GGTACCCTCCGCGTGGTCGTACATG-3′;Ri-ROSES1R:5′-GGATCCCTATGTCCCACTCCCAGGGTTT-3′.扩增程序为95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火30 s,72 ℃延伸15 s,循环40次;扩增后,65 ℃退火5 s;95 ℃溶解5 s(每次循环增加0.5 ℃,循环60次)进行溶解曲线分析.实时PCR在Bio-Rad CFX 96上运行,数据用CFX Manager 2.0软件进行分析.
1.1. 材料与试剂
1.2. OsROSES1系统进化树的构建
1.3. OsROSES1基因RNAi载体构建
1.4. 转化野生型植株获得干涉植株及鉴定
1.5. OsROSES1基因RNAi转基因植株表型分析
1.6. OsROSES1基因表达分析
-
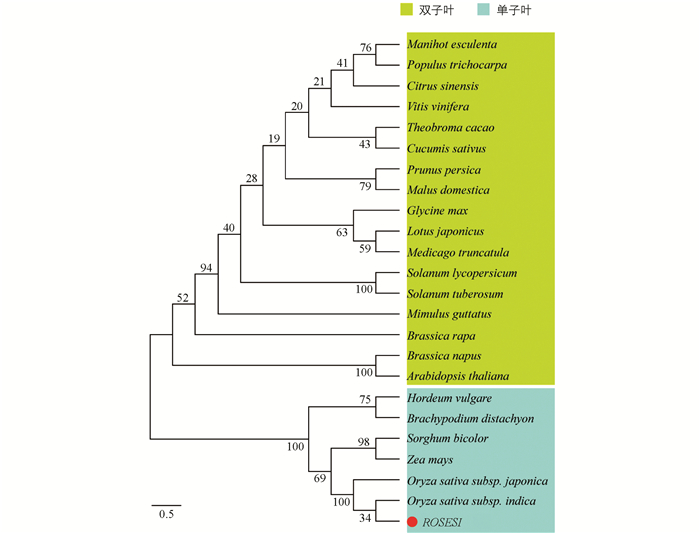
结构域分析结果表明,ROSES1蛋白含有779个氨基酸碱基,编码包含一段植物特定的POX结构域的BEL1-Like“同源盒基因”转录因子.进化树分析表明这些蛋白细分为单子叶和双子叶2个主要分枝,具有高度的保守性(图 1).在蛋白结构域鉴定的基础上,同源结构域和POX域存在于所有单子叶和双子叶植物的ROSES1同源染色体上,表明ROSES1蛋白可能在单子叶和双子叶分枝之前出现.
-
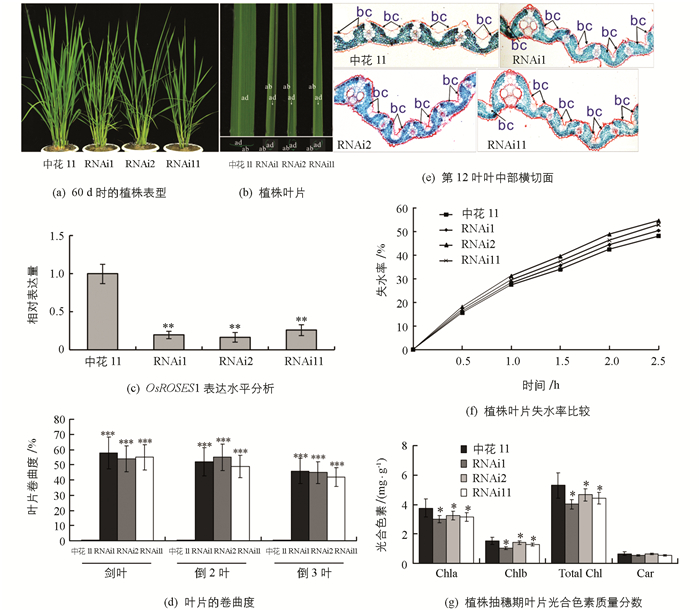
利用RNAi技术进一步确认OsROSES1基因功能,构建RNAi载体pUbi:ROSES1-RNAi并转化到野生型中花11(Oryza sativa ssp. japonica)中,获得19株转基因植株,其中阳性植株分蘖期开始表现为植株整体变矮,叶片变短且内卷(图 2a和b).与中花11相比较,阳性干涉转基因植株(RNAi1,RNAi2和RNAi11)的OsROSES1表达水平显著降低(图 2c);干涉转基因植株叶片后3叶(剑叶、倒2叶和倒3叶)的卷曲度分别为56%,52%和44%(图 2d);石蜡切片显示干涉转基因植株叶片卷曲部位泡状细胞明显变小(图 2e).生长期60 d时,对气孔密度增加的转基因植株叶片取样在常温条件下每隔0.5 h称量叶片鲜质量1次,一直到2.5 h,然后进行失水率分析(图 2f),结果表明干涉转基因植株下调了OsROSES1,显著增加了叶片气孔导度,引起叶片失水速度加快.进一步检测抽穗期干涉转基因植株光合色素质量分数,结果与中花11相比,3个干涉转基因植株光合色素质量分数显著降低(图 2g).
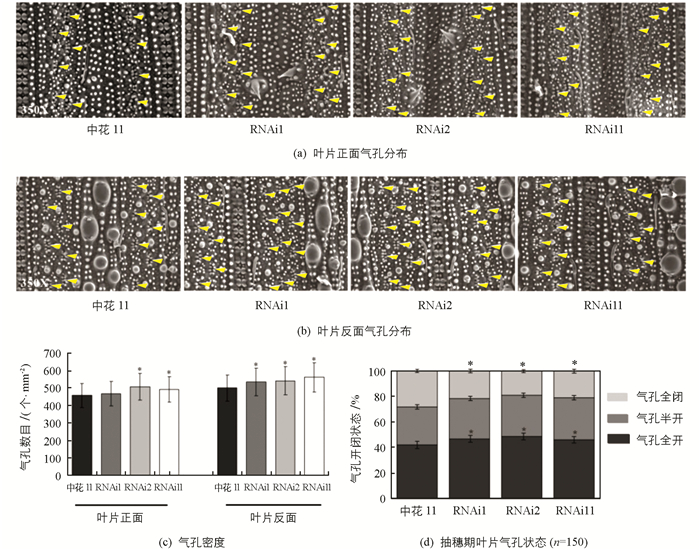
为了验证OsROSES1基因是否调控水稻气孔发育和运动,通过电镜扫描60 d大小的中花11和3个干涉转基因植株叶片气孔,观察比较气孔密度和开度.叶片上下表面气孔的数量和分布如图 3a至图 3c,每张叶片反面气孔数目比叶片正面气孔数目多.相比于中花11,干涉转基因植株RNAi1,RNAi2和RNAi11的正面叶片单位面积气孔数目分别增加了7.72%,10.91%和7.29%,反面叶片单位面积气孔数目分别增加了12.68%,14.32%和12.32%.抽穗期对中花11和3个干涉转基因植株叶片气孔全开、半开和全闭3种状态的数量进行了统计,结果显示,RNAi1,RNAi2和RNAi11气孔全开状态百分比分别为47.4%,48.8%和46.2%,显著高于中花11的42.2%,而RNAi1,RNAi2和RNAi11气孔全闭状态百分比分别为22.2%,19.2%和21.4%,显著低于中花11的28.1%(图 3d).干涉转基因植株下调了OsROSES1,导致叶片气孔密度和开度显著增加.
在成熟期对干涉转基因植株农艺性状调查结果表明,与中花11相比较,阳性干涉转基因植株(RNAi1,RNAi2和RNAi11)的株高、有效穗数和千粒质量显著降低,结实率极显著降低(表 1).由于OsROSES1功能的缺失影响了细胞增殖和扩展,使器官细胞数目变少和细胞大小变小,植株表现出株高变矮、有效穗数减少、结实率和千粒质量降低.
-
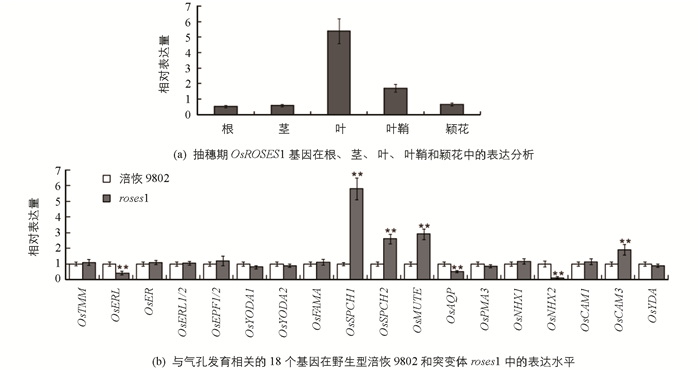
实时定量PCR分析结果表明,OsROSES1基因在所有器官中表达,其中在幼嫩的叶片和叶鞘中表达水平较高(图 4a).用以调控气孔密度和开度相关基因、保卫细胞离子通道调控基因、液泡膜Na+/K+逆向转运蛋白及水通道蛋白等相关基因对野生型涪恢9802和roses1突变体进行定量表达分析,结果表明,与野生型涪恢9802相比较,roses1突变体正调控表皮细胞不对称分裂,增加叶片气孔密度和开度的调控因子OsSPCH1,OsSPCH2,OsMUTE和保卫细胞离子通道蛋白OsCAM3表达量极显著升高,而抑制表皮细胞不对称分裂的调节因子OsERL,液泡膜Na+/K+逆向转运蛋白OsNHX2和水通道蛋白OsAQP表达量极显著降低(图 4b,表 2),该结果与植株整体大小变小、叶片变短且内卷、叶片气孔密度和开度增加、叶片蒸腾速率加快引起叶片卷曲的干涉阳性转基因植株表型一致.
2.1. OsROSES1基因进化树分析
2.2. 转基因RNAi植株的获得及表型分析
2.3. OsROSES1基因的表达分析
-
植物成熟器官的形成需要经历两个过程:首先通过紧密协调的细胞生长与细胞分裂实现细胞增殖,随后转为伴随着核内复制的细胞扩展与分化[25-26].这两个过程是连续进行的,许多基因能通过转录调节、蛋白酶体合成或降解、植物激素调节或松弛细胞壁、小RNA调控等途径作用于这两个过程调控器官的大小. OsROSES1基因编码包含一段植物特定的POX结构域的BEL1-Like“同源盒基因”,在水稻根或顶端分生组织、居间分生组织和维管束细胞组织部位高表达,并参与调控顶端生长和侧生器官原基的细胞分裂和细胞扩展过程[15].本文利用RNA干涉下调OsROSES1的表达,阳性转基因植株从分蘖期开始,植株逐渐变矮,叶片变短且内卷,光合色素质量分数显著下降.叶片卷曲部位泡状细胞明显变小,单位面积气孔数目和气孔导度增加,蒸腾速率加快使叶片呈现卷曲;分蘖期叶片光合色素质量分数显著下降,导致植株成熟期株高变矮、有效穗数减少、结实率和千粒质量降低.表明OsROSES1通过调控细胞增殖和扩张控制器官大小,改变叶片气孔密度和开度,从而影响光合作用.
对于水稻器官大小的调控,目前仅报道了WUS,OSH1,OSH15等少数几个调控因子,WUS是水稻形成分蘖(顶端分生)初始化分生组织非常重要的同源转录因子[27].同时,水稻WUSCHEL-RELATED HOMEOBOX(WOX)基因被证明是明显促进根的顶端分生组织和冠根的形成[28].同样,水稻ORYZA SATIVA HOMEOBOX1(OSH1)和KNOTTED-LIKE HOMEOBOX[29]基因被证明促进顶端分生组织处于未分化状态[30].水稻osh1突变体发芽之后表现为促进顶端分生组织的形成.水稻osh1和d6(编码同源框蛋白OSH15)基因双突变体表现为顶端分生组织不发达,结果表明在水稻胚胎形成期间需要这2个基因激活或促进顶端分生组织形成[30].本研究的ROSES1蛋白属于同源框亚科,包含2个功能域:1个DNA同源结构域和1个植物特有未知功能POX结构域[31].水稻WUS同源基因和其他同源框基因对顶端分生组织、根的分生组织的萌发和腋生器官的原始形成起到重要调控作用,与之不同的是,OsROSES1是同时在胚胎形成期间启动初生分生组织和所有腋生器官及组织的次生分生组织,如侧根、分蘖、花穗分枝、小穗原基和分别在茎周围和叶主脉两侧的维管束[15],但是,OsROSES1是如何协同WUS和其他同源框基因调控分生组织的形成需要进一步研究.
水稻roses1突变体器官大小的变小影响叶片气孔的密度和开度,蒸腾速率加快使叶片呈现卷曲,引起叶片光合色素质量分数显著下降,导致水稻roses1突变体叶片发生早衰,表明OsROSES1可能通过调控下游的一些目标基因从而调控气孔密度和开度[15].类似的研究表明,一个属于Ⅳ级同源框—亮氨酸链家族的同源框START转录因子的激活表达,使气孔的密度减少提高其抗旱能力[32].作为植物气孔密度和开度的正调控因子,b HLH转录因子SPCH促进拟分生组织母细胞的形成,调控起始不等分裂、扩增不等分裂和间隔不等分裂[33-34],而MUTE终止拟分生组织细胞的干细胞活性并促进其分化为保卫母细胞[33]. ERECTA-LIKE(ERL)通过抑制保卫母细胞的分化负调控气孔发育,在叶表皮气孔细胞中广泛表达[35]. NHX(Na+,K+/H+反向转运体)主要负责将胞质中多余的Na+和K+储存在液泡里,同时将H+从液泡排到胞质中,拟南芥气孔开启依赖NHX1和NHX2在保卫细胞液泡积累K+的过程[36].钙调蛋白作为钙离子的主要感受器,以Ca2+信使途径参与了干旱和ABA引起的地上部叶片气孔开闭的胞间信号传导过程[37].水通道蛋白(aquaporin,AQP)参与调节叶肉细胞内二氧化碳的运输,影响植物叶片气孔运动及光合作用[38].本研究中,与野生型涪恢9802相比较,roses1突变体正调控表皮细胞不对称分裂,增加叶片气孔密度和开度的调控因子OsSPCH1,OsSPCH2,OsMUTE和保卫细胞离子通道蛋白OsCAM3表达量极显著升高,而抑制表皮细胞不对称分裂的调节因子OsERL、调控叶片气孔开度的质膜上的H+泵家族基因OsNHX2和水通道蛋白OsAQP表达量极显著降低.该结果表明OsROSES1可能直接或间接调控了气孔的密度和开度以及气孔运动涉及囊泡运输、液泡融合、膜H+泵和离子平衡蛋白. OsROSES1是如何调控水稻器官发育还需要进一步研究.
-
本研究利用RNA干涉下调OsROSES1的表达,分蘖期植株整体变矮,叶片变短且内卷,卷曲部位泡状细胞明显变小,增加了叶片单位面积气孔数目,由于蒸腾速率加快引起叶片卷曲,从而显著降低了植株光合色素质量分数,导致水稻roses1突变体叶片发生早衰,成熟期植株表现出株高变矮、有效穗数减少、结实率和千粒质量降低,该结果进一步表明ROSES1可作为水稻BEL1-Like“同源盒基因”,在植物器官发育过程中通过调控细胞增殖和扩张从而控制器官大小和光合作用.这为下一步研究OsROSES1在水稻器官发育调控网络中的功能奠定了基础.



 下载:
下载: