-
藜麦(Chenopodium quinoa Willd),一年生藜科谷物,又称南美藜、印第安麦、奎藜、奎奴亚藜等,原产于南美洲安第斯山区,至今已有5 000~7 000年的种植和食用历史,在玻利维亚、智利等国家一直是当地居民最主要的粮食,适合生长在高海拔、少雨、空气稀薄、寒冷、日照强、土壤贫瘠的沙质甚至碱性地区,这些地区一般作物较难生长,环境污染很低[1].众多研究者认为,无论在植物界还是动物界都没有一种食物能像藜麦那样提供全面平衡的人体所需营养元素[2].藜麦有极高的营养价值,平均含蛋白质16.7%、脂肪7.0%、纤维素1.39%、全钙0.74%、全铁0.73%、全磷0.43%.除含有丰富的钙、磷和铁,还含有丰富的维生素E和一些B族维生素[3]. 20世纪90年代之后,人们开始认识到藜麦的营养价值和它的发展潜力,欧洲国家和美国、加拿大相继进行了引种和栽培.联合国大会将2013年定为“国际藜麦年”,旨在让世界关注藜麦的生物多样性和营养价值在提供粮食和营养安全、消除贫困以及在支持实现千年发展目标等方面所能发挥的作用[4].近年来,随着藜麦独特的营养价值被人们发现,在国际市场身价倍增,被誉为“粮食之母”而风靡全球,成为时尚高端的营养食品,美国宇航局甚至将藜麦选定为太空粮食[5-6].
1996年,西藏自治区农作物品种审定委员会审定出合格品种21个,其中3个为藜麦引进驯化品种.自2014年开始,藜麦在西藏进行了推广,近2年的推广面积均在667 hm2左右,推广面积较大的地区分别为日喀则、林芝和拉萨.从农牧民手中回收价格一般为24~30元/kg,加工后售价可达200~300元/kg,大大增加了当地农牧民种植积极性和经济收入.
由于藜麦突出的营养品质及抗旱性、耐寒性,许多人对其进行了化学组成、营养价值、生物学特性、栽培技术、病害、苗期抗霜冻能力等方面的研究[7-8].尽管对藜属植物的染色体数已进行过一定研究[9-11],但至今依然缺乏对其染色体核型的详细分析[12].染色体是遗传物质的载体,植物染色体制片和核型分析技术是细胞遗传学中最基本、最常用的方法,在物种亲缘关系鉴定、染色体变异、杂种分析等方面得到广泛应用[13].关于藜麦细胞学方面的研究已有一些报道,但由于藜麦植物染色体较小使得核型分析的难度增大,目前只有少量研究报道了其染色体数目,对核型的研究极少,更没有人对西藏目前主推的藜麦品种(品系)进行深层次研究.因此,本实验采用不同处理方法进行藜麦核型分析,以期筛选出藜麦染色体制片的最适处理条件,探明其染色体数目及核型,为藜麦遗传育种工作等提供细胞学数据.
HTML
-
2016年在西藏农牧学院种植的、代号为W4(当前生产表现良好)的藜麦(Chenopodium quinoa Willd)种子,由西藏农牧学院植物科学学院旺姆教授提供.
-
将藜麦种子放入烧杯,用蒸馏水清洗去掉杂质及种子表层的水溶性皂角苷,浸泡10 min左右,置于垫有两层滤纸的培养皿中,放入温度23 ℃、湿度72%、无光照的生化培养箱中发根,期间注意保持培养皿中的湿度,当根长至1~2 cm时,于上午09:00-11:00之间取材.预处理设置不同的处理方法及处理时间(表 1),以探讨最佳的藜麦根尖预处理方法.
预处理之后,用新配制的卡诺固定液(无水乙醇:冰乙酸=3:1)进行固定(24 h),然后依次用95%,85%,75%乙醇冲洗根尖,最终置于70%~75%乙醇中,(4~5) ℃冰箱保存.
-
处理好的根尖用蒸馏水清洗2~3遍,再用酒精冲洗干净,控净水份,加入1 mol/L HCl、置入60 ℃预热好的恒温水浴锅中解离5~20 min,每分钟取样1次,将材料用蒸馏水清洗干净,取根尖分生区1~2 mm,使两片载玻片呈十字交叉法压片后打开,在每片载玻片上各滴加1小滴卡宝品红染色液染色3 min.将载玻片放在平整的桌面上,盖上盖玻片,盖玻片用左手手指压紧,防止盖玻片滑动,再用铅笔头垂直轻轻敲打盖玻片,使材料均匀分散后压片.
-
将准备好的制片放在OLYMPUS CX41显微镜下观察,并选择分散较理想的中期染色体图像进行显微摄影.按照李懋学等[14]的核型分析标准:统计的细胞数目应在30个以上,其中85%以上的细胞具恒定一致的染色体数,即可认为是该植物的染色体数目.将图片输入电脑中分别用Scopephoto和ImageJ软件进行测量,利用Excel计算臂比、着丝粒指数、核型不对称指数、染色体长度比等核型参数;有随体的染色体,其随体长度及次缢痕长度未计入全长.核型分类以Stebbins[15]方法进行,并按照乔永刚等[16]的方法利用染色体相对长度的参数在Microsoft Office Excel中绘出核型模式图.利用Adobe Photoshop及Excel制作核型图和核型模式图.
1.1. 材料
1.2. 方法
1.2.1. 根尖材料的获取
1.2.2. 染色体制片
1.2.3. 镜检及核型分析
-
每种植物的根尖染色体大小及数目不同,不同的预处理方式对同一植物的处理效果也不尽相同,因此不同植物有不同的最佳预处理方式.用0.002 M的8-羟基喹啉分别处理3~8 h,镜检观测结果表明:在低于6 h的处理中,染色体前期分裂相较多,能辩清染色体数目,但主缢痕不易辨明.处理时间超过6 h后,染色体弯曲、拉长,边缘模糊.因此,在非离体情况下用0.002 mol/L的8-羟基喹啉处理6 h为最佳处理时间.用0.05%秋水仙素溶液非离体处理3~6 h时镜检显示:总体上染色体分散程度不够,虽能辩清染色体数目,但主缢痕不易辨明.处理5~6 h与处理3~4 h相比,染色体结构相对清晰,因此长时间、低秋水仙素溶液浓度处理相对较好.冰冻处理法主要存在问题为主缢痕不清晰,无法很好地辨明染色体结构.用0.1%秋水仙素处理离体根尖,处理3 h与处理2 h相比,染色体结构更为清楚,主缢痕清晰可辨.因此,在以上4种处理中,0.1%秋水仙素在5 ℃下、离体预处理根尖3 h效果最佳.
-
用1 mol/L HCl、恒温水浴锅预热至60 ℃进行酸解.酸解时间设置为5~20 min,每分钟取样1次,每个处理选取5张制片进行观察.结果显示:当酸解时间低于12 min时,根尖一般较硬,在后期压片时材料不易分散,细胞重叠影响对染色体形态和数目的观察.当酸解时间超过15 min后,制片中染色体染色较浅,观察效果仍然欠佳.酸解13~14 min时细胞易分散且染色体着色较深,易于观察,因此酸解13~14 min为最佳酸解时间.在此过程中还发现,用固定液固定24 h后立即进行酸解和制片,酸解需要时间相对要短一些,可以在12 min左右,而且染色体形态及数目相对清晰.如果固定时间相对较长,则酸解需要的时间也要长一些,但一般不超过15 min.这个结果说明固定后立即进行酸解和制片,其观察效果比在70%~75%乙醇中保存一段时间的材料要好.
-
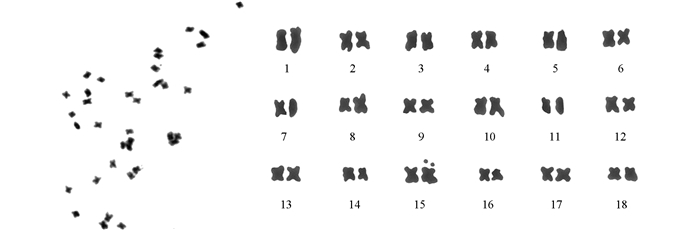
对根尖染色体制片进行镜检,并选择50个染色体清晰而且分散良好的中期分裂相观察计数,所有观察统计的细胞染色体数目均为2n=36,这个结果与文献[17-18]的结果一致.未发现非整倍体细胞,具体染色体分裂相及核型见图 1.
-
各选取5张比较清晰的藜麦染色体中期分裂相图片进行核型分析,确定W4的染色体数目为2n=36.在本实验中,最长染色体与最短染色体的比值为1.85:1.臂比值大于2:1的染色体占全部染色体的11.11%,属于2B型.核型不对称系数(长臂总长/全组染色体总长)为57.87%.在藜麦的18对染色体中,16对为中部着丝点染色体(m),占总染色体数的88.89%;2对为近中部着丝点染色体(sm),占总染色体数的11.11%.核型公式为2n=36=32 m(2SAT)+4 sm.具体核型参数见表 2,核型模式见图 2.
2.1. 藜麦染色体制片方法优化
2.1.1. 预处理方法优化
2.1.2. 酸解时间优化
2.2. 藜麦染色体数目
2.3. 藜麦染色体核型参数
-
陈于和等[19]曾用豌豆根尖做过离体与非离体的处理比较,认为在离体条件下药剂和低温预处理的中期分裂指数有所提高,且非离体条件的中期分裂指数要高于离体条件的中期分裂指数.本实验中离体处理效果却好于非离体处理,但因为本实验中秋水仙素溶液浓度不同,因此没有确切的比较标准,今后可对这个进行比较.藜麦根尖很小,离体后容易在清洗过程中丢失,且离体步骤相对耗时较多,非离体处理更利于节约实验时间成本.
有研究表明,对同一物种根尖有丝分裂进行阻断的方法和时间不同会产生不同凝缩程度的中期染色体,并导致染色体长度产生较大差异[20].因此,在对西藏其他藜麦品种(品系)核型进行研究时,应采用同一处理方法进行比较,应当制定一套其核型分析步骤的标准,这样既方便进行同一个属或种内的核型比较,也方便学术交流.
实验中曾一度出现次缢痕无法观察到或无法看清的状况,主要原因在于藜麦染色体较小(未发现非整倍体细胞也可能与此有关).但在排除制片的操作问题及提升显微镜规格后,对目标的观察效果有较大改观.
此次实验仅采用了1个西藏的藜麦品系,今后应对更多藜麦品种(品系)核型进行探讨,以期对藜麦遗传育种研究提供更多、更详实的细胞学数据.






 DownLoad:
DownLoad: