-
开放科学(资源服务)标志码(OSID):

-
BR缺陷突变体如拟南芥中的DET2,DWARF4(DWF4)和CPD,番茄中的DWARF,豆科植物中的LKB;BR不敏感突变体如拟南芥中的DWF12,BRI1,BIN2,BAK1,豆科植物中的LKA,番茄中的CURL-3等都不同程度表现为根长、株高、花序、角果、叶柄、胚轴的减短、顶端优势的减弱、育性衰减、开花和叶片卷曲、维管束和光形态建成上的变化,显示BR在植株生长和发育中的重要作用[1]. 通过在植株中超表达DWF4基因,可以使植株更高,叶片更多[2]. 目前还发现BR和其他激素在侧根发生方面起到的协同作用[3],然而对于下胚轴和根的向地性方面起到的却是反作用. 低浓度水平下的BR促进根的伸长,高浓度则抑制根的伸长[3]. 多种实验结果证明BR能促进产量提高. 水稻BR缺陷突变体BRD2致使谷物颗粒变短而且缩小,而DWF11突变体也呈现出种子减少现象[4]. 相反,超表达BR生物合成基因将增大水稻颗粒,增加水稻产量,转基因水稻与野生型相比,分蘖数更多[5]. 在拟南芥中,BR对种子发育有决定性作用,包括种子大小、种子形状和种子数量等[6]. 拟南芥BR缺陷突变体DWF5结出的种子变小[7],BR缺陷突变体DET2和BRI1-5的种子也都比野生型小,而且DET2和BRI1-5的种子质量比野生型分别轻17%和10%,BZR1功能获得型突变体BZR1-1D的种子与野生型相比,不仅更小而且变得更轻[6]. 在拟南芥中,超表达DWF4可以提高种子数量. 就如Choe等[2]研究过表达BR合成关键基因,获得了转基因拟南芥植株,对该转基因植株的果荚和分枝数统计调查显示其数量均增加2倍,种子产量增加了59%,GC-MS分析表明该转基因植株內源BR水平确实有所升高. 另外,BR还调节植物花期和衰老. BR相关基因CPD,可以通过光周期途径调节开花,而CPD同系物GmCPDs促成开花发育,调控花期[8]. 水稻BR信号途径相关基因SDG725,也和花期调控相关[9].
目前,在BR的生物合成、代谢作用、信号转导途径中都有很多报道,其中DWF4编码细胞色素P450酶,与CPD基因处于同一家族不同亚族[10]. DWF4的mRNA水平因外源施加油菜素内酯(brassinolide,BL)而大幅下降,因施加BRZ(油菜素内酯抑制剂)而大幅提高,证实DWF4在BR平衡调节上起到了重要的作用[11]. DWF4敲除突变体的内源BRs缺失将致使植株矮小,突变体植株细胞伸长受到限制[12]. 对野生型植株施加BRZ,将抑制或结合DWF4酶[13],也会造成内源BRs缺失致使植株矮小. DWF4是维持BR平衡的关键基因,因其可以迅速而准确地对内源BR水平响应表达变化[14]. 本实验中通过农杆菌介导转化法将DWF4基因导入芥菜中,研究DWF4基因在芥菜生长过程中的作用与功能,为解析DWF4基因参与BR信号传导的机制提供直接或者间接的实验证据,也为今后开发生物技术手段调控植物生长发育奠定理论基础.
HTML
-
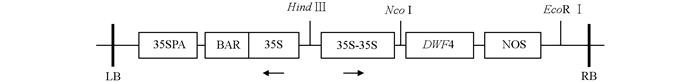
含pCABarDWF4植物表达载体质粒的农杆菌由西南大学蔬菜学实验室提供,表达载体结构示意图见图 1.
-
利用农杆菌介导法转化芥菜,芥菜种子所需消毒时间为75%的乙醇消毒1 min,0.1%的升汞处理8 min,所需农杆菌侵染时间为8 min,使用的预培养基成分为MS+2 mg/L 6-BA(6-苄基腺嘌呤)+0.2 mg/L NAA(萘乙酸);筛选培养基成分为MS+2 mg/L 6-BA+0.2 mg/L NAA+8 mg/L PPT(膦丝菌素)+400 mg/L Carb(羧苄青霉素);生根培养基成分为MS+0.2 mg/L NAA+8 mg/L PPT+400 mg/L Carb.
-
采用CTAB法提取获得的转基因芥菜植株基因组DNA进行PCR扩增. 引物DWF4-1和DWF4-2用于pCABarDWF4转基因芥菜植株DWF4基因片段的检测. PCR扩增参数:95 ℃预变性5 min;95 ℃ 40 s;56 ℃ 40 s;72 ℃延伸2 min;30个循环;72 ℃延伸10 min. 获得的扩增产物经1%琼脂糖凝胶电泳检测.
-
用TIANGEN RNA prep pure Plant Kit提取试剂盒提取pCABarDWF4转基因芥菜植株总RNA,根据TransScript First-Strand cDNA Synthesis SuperMix(TaKaRa)试剂盒说明书进行cDNA合成,获得的第一链cDNA稀释5倍后作为RT-PCR模板. 以芥菜BjActin为内参,对转基因植株和非转基因植株中的DWF4基因BR生物合成结构基因的表达情况进行Real Time RT-PCR分析,反应在C1000/CFX96仪器(Bio-Rad,美国)上完成,具体操作按照SYBR© Premix Ex TaqTM Ⅱ说明书进行. 利用Bio-Rad CFX96荧光定量PCR仪CFX Manager Software软件(2-ΔΔCT法)和Microsoft Excel软件分析数据.
-
pCABarDWF4转基因芥菜植株及非转基因植株种子播种于培养皿中,铺上滤纸,只施加蒸馏水,置于25 ℃培养室中,光照条件和黑暗条件下培养4 d,每个基因型进行3次重复,每个重复20粒种子,最后调查其根长、下胚轴长、总长,在滤纸上吸干水后测其鲜质量,最后置于60 ℃烘箱中烘干测其干质量. pCABarDWF4转基因芥菜植株及非转基因植株播种于培养盆中,播种后每隔15 d测量其株高、开展度、单株最大叶长和最大叶宽,自花授粉结束后测量主枝长度、主枝果荚数、主枝种子数、一次分枝数、一次分枝果荚数、一次分枝种子数、二次分枝数、二次分枝果荚数、二次分枝种子数并测量千粒质量和籽粒密度. 千粒质量的测定:转基因植株和野生型植株种子分别取100粒称质量,3次重复,取其平均值. 籽粒密度(D)的测定:取200粒称质量记为M,然后将籽粒放入事先盛有定量体积V1酒精的量筒中,然后读取体积V2,则籽粒体积V=V2-V1. 籽粒密度D=M/V,单位g/mL,3次重复.
1.1. 材料
1.2. 芥菜遗传转化
1.3. 转基因芥菜植株的PCR鉴定
1.4. pCABarDWF4转基因芥菜植株DWF4基因、BR生物合成结构基因、转录调控基因的表达分析
1.5. pCABarDWF4转基因芥菜植株形态分析
-
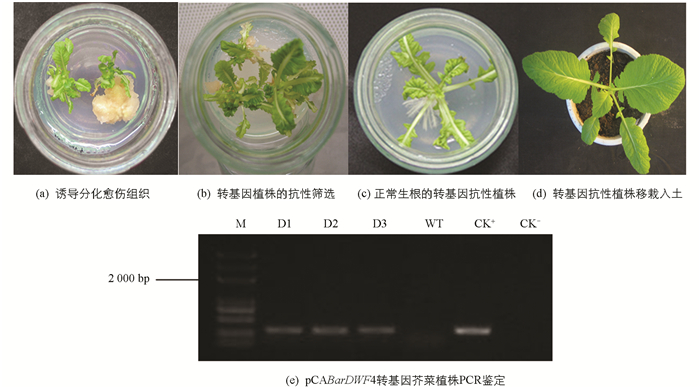
PCABarDWF4表达载体通过农杆菌介导的下胚轴转化,侵染后的下胚轴转接至含8 mg/L PPT和400 mg/L Carb的抑菌筛选培养基上,对获得的不定芽进行多代筛选,淘汰未转化的白化芽和嵌合体,获得多个具PPT抗性的绿色愈伤组织和芽(图 2a和图 2b),抗性芽接种至生根培养基上进行生根培养(图 2c). 抗性植株炼苗后,移栽至泥炭土营养钵中(图 2d),最后定植于大田中,正常管理至植株开花. 提取pCABarDWF4转基因芥菜植株T0代基因组DNA从3株转基因植株中扩增到预期大小300 bp左右的目的片段(图 2e).
-
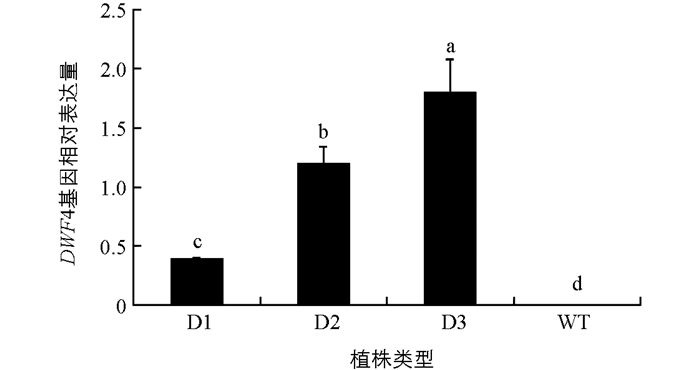
提取pCABarDWF4转基因芥菜植株的RNA,以芥菜BjActin为内参进行DWF4基因的定量表达分析. 从结果(图 3)可以看出,pCABarDWF4转基因芥菜D1,D2和D3植株中,DWF4基因均有不同程度的表达,但表达量在植株之间有差异,D2和D3植株中目的基因表达量较高,对照野生型植株(WT)未检测到明显的DWF4表达.
-
将pCABarDWF4转基因芥菜种子进行光照处理后调查其生长状况. 从表 1可以看出,D2和D3的根长显著高于野生型,D1的根长虽然高于野生型但未达到统计学意义. D1和D3的下胚轴长显著高于野生型. D1,D2和D3苗期总长均显著高于野生型. 在鲜质量方面,转基因植株以及野生型植株之间差异无统计学意义. 在干质量方面,D1和D3显著高于野生型,D2的干质量虽然高于野生型但差异无统计学意义. 相比之下,苗期生长最为旺盛的是D3. 综上所述,在光照条件下,在芥菜中超表达DWF4基因能一定程度上促进苗期生长.
与光照条件下相比,在黑暗中,不管是转基因植株还是野生型植株,苗期下胚轴都大幅度伸长,但植株纤弱,子叶黄化. 从表 2中可以看出,D2和D3的根长显著高于野生型,D1的根长虽然高于野生型但差异无统计学意义. D1和D3苗期下胚轴长均显著高于野生型,D2下胚轴长低于野生型. 在苗期总长上,D1和D3显著高于野生型,由于D2下胚长较短的原因,所以其总长与野生型差异无统计学意义. 在鲜质量和干质量上,转基因植株与野生型植株之间差异无统计学意义. 从实验结果可以看出,在黑暗条件下,DWF4基因能在一定程度上促进苗期根和下胚轴伸长.
光照条件与黑暗条件下不同的是,光照条件下苗期干质量差异有统计学意义. 不论是置于光照还是黑暗条件下,D3生长都是最为旺盛的,苗期总长和下胚轴长都显著高于野生型. 通过对转基因植株苗期光照和黑暗条件下生长情况的统计,可以发现转基因植株根和下胚轴长一定程度上都高于野生型植株,而且D3表现最好. 结合D3转基因株系DWF4基因较高的表达水平,显示DWF4基因表达较高者,其苗期根以及下胚轴生长速度会更快.
-
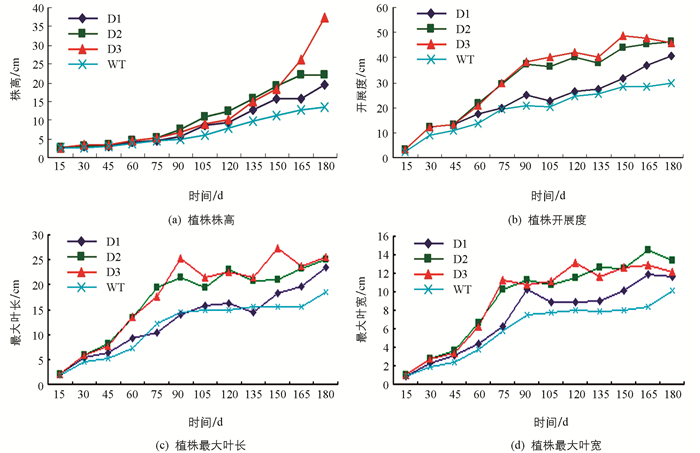
将pCABarDWF4转基因芥菜植株置于人工气候室内培养观察,从其整个生命周期中,转基因植株比野生型植株生长更加旺盛. 在60 d时,转基因植株就已经比野生型植株长得更壮,其叶片伸展更开(图 4a).
到180 d时,转基因植株开始逐渐抽薹,而野生型植株还没有抽薹(图 4c). 转基因植株最早开花时间比野生型植株提前一个多月. 转基因植株和野生型植株花器官相比,转基因植株的花瓣较大(图 4d). 对比转基因植株和野生型植株同时期的叶片,可以发现转基因植株叶片伸展更长、更宽,而且叶片颜色更加浓绿(图 4b). 从图 5中可以发现,随着时间的延长,转基因植株和野生型植株的株高、开展度、最大叶长和最大叶宽也随着增长,但是转基因植株生长明显快于野生型植株. 转基因植株之间相比,D2和D3生长势明显好于D1.
-
进一步对转基因植株结荚结籽情况进行了调查. 转基因植株枝条上比野生型植株结有更多的种子(图 6a),转基因植株果荚长度高于野生型植株果荚长度(图 6b),且转基因植株果荚结籽数更多(图 6c和图 6d).
从表 3中可以看出,转基因植株主枝长度远远超过野生型植株. 除D3外,主枝平均果荚数也超过了野生型,转基因植株主枝果荚长度和所结种子数均高于野生型. 转基因植株一次分枝数和二次分枝数高于野生型,且一次分枝果荚数、一次分枝所结种子数和二次分枝果荚数、二次分枝所结种子数也都高于野生型. 综合来看,转基因植株种荚率、全株总种荚数和所结总种子数均高于野生型植株. 对千粒质量和籽粒密度测量数据来看,D1和D2超过野生型而D3与野生型基本一致. 结果显示,在芥菜中超表达DWF4基因具有明显增加种子产量的作用.
-
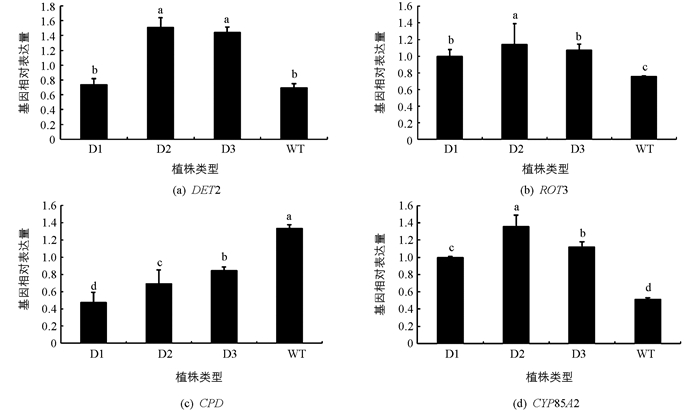
DET2,ROT3,CPD,CYP85A2等基因被认为参与了BR的生物合成. 在pCABarDWF4转基因植株中,DWF4下游基因DET2在D2和D3株系中平均表达量是野生型的2倍,说明超表达DWF4基因促进了下游DET2基因的表达(图 7a).
DET2基因下游的ROT3基因在转基因植株中的平均表达量高出野生型植株的40%(图 7b).
CPD基因代谢油菜甾醇(campesterol)和其他中间产物,而DWF4基因也参与代谢油菜甾醇,DWF4基因与CPD基因可能存在竞争作用. 在本实验中,pCABarDWF4转基因芥菜植株的CPD基因表达量都低于野生型,其平均表达量只达到野生型植株表达量的一半,说明DWF4基因与CPD基因表达呈负相关(图 7c).
CYP85A2是BR生物合成途径中的重要因子,在pCABarDWF4转基因芥菜植株中,他们的相对表达量都不同程度高于野生型,CYP85A2是在BR合成途径中最后一步反应的基因,说明转基因pCABarDWF4芥菜中,超表达DWF4基因促进了下游相关BR合成基因的表达,促进了BR的生物合成.
-
BR生物合成除了结构基因的表达外,还受到细胞内其他转录激活或者转录抑制基因的调控.
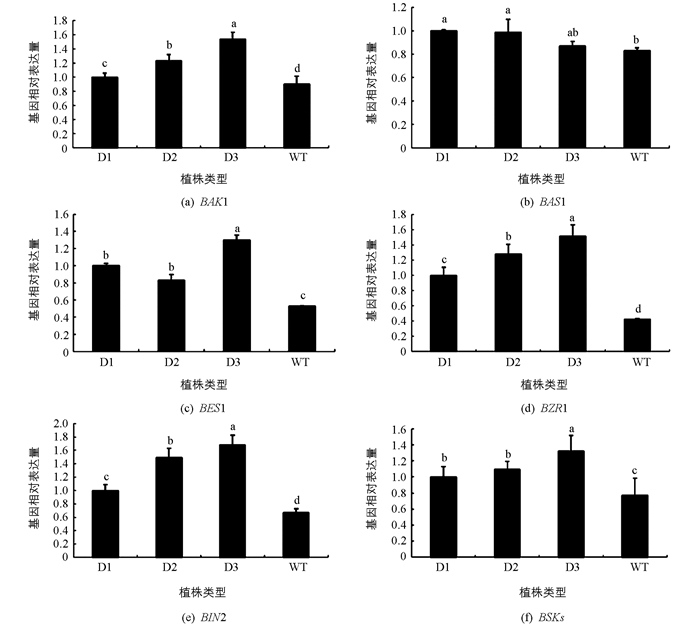
BAK1是一类重要的类受体蛋白,参与调控BR的合成,调节植物生长发育. 在pCABarDWF4转基因植株中,BAK1基因表达量均比野生型植株表达量升高,且平均值高出近40%(图 8a).
BAS1基因表达被外源BR诱导,功能应该是维系内源BR的恒定水平,其羟化C26,影响植株体内BR量,是一个油菜素内酯失活基因. 在本研究中,虽然转基因植株中BAS1的平均表达量0.95高于野生型植株表达量0.83,但随着DWF4基因表达水平上升,BAS1基因表达水平降低(图 8b).
BES1和BZR1是油菜素甾醇信号途径下游最重要的一类转录因子,参与油菜素甾醇合成转录的诱导和抑制调控. 在本研究中,转基因株系中BES1和BZR1的平均表达量分别是野生型植株的2~3倍(图 8c和图 8d),说明超表达DWF4基因导致细胞内BR积累水平上升后,引起BES1和BZR1基因表达水平上升,促进细胞内积累更多的BR.
BIN2在BR途径中起到调控BES1和BZR1的作用,在转基因植株中BIN2基因的平均表达量是野生型植株的2倍(图 8e),BES1和BZR1表达量较高,那么BIN2表达量应和这两个基因表达量相一致.
BSKs是一类参与油菜素内酯信号转导的植物特异受体类胞质激酶,参与BR信号转导途径,在转基因植株中其平均表达量高于野生型植株近50%(图 8f).
BAK1,BAS1,BES1,BZR1,BIN2,BSKs都参与了BR合成的转录调控,调节内源BR水平,在pCABarDWF4转基因芥菜植株中,随着DWF4基因表达水平的提高,大多数调控基因BAK1,BZR1,BIN2和BSKs的表达量与DWF4表达量呈正相关,出现明显上调. 而维系细胞内内源BR稳定水平的BAS1基因,则在不同植株之间的表达水平变化小.
2.1. pCABarDWF4转基因芥菜植株的获得及鉴定
2.2. pCABarDWF4转基因芥菜植株DWF4基因的表达分析
2.3. pCABarDWF4转基因芥菜植株苗期光暗处理表现分析
2.4. pCABarDWF4转基因芥菜植株生长情况分析
2.5. pCABarDWF4转基因芥菜植株结籽情况分析
2.6. pCABarDWF4转基因芥菜植株BR生物合成结构基因
2.7. pCABarDWF4转基因芥菜植株BR转录调控相关基因表达分析
-
DWF4编码C22羟基化酶,其转录水平受BR反馈抑制,其可以迅速而准确地对内源BR水平响应表达变化[11, 14],是BR合成关键基因之一. 在拟南芥中,不论是光照还是黑暗条件下,通过超表达DWF4基因,拟南芥幼苗下胚轴长度远远高于对照野生型[2],本实验中,在芥菜中超量表达DWF4基因后,与野生型相比,转基因苗期根茎伸长变快,与在拟南芥中的实验结果一致. 在拟南芥和烟草中超表达DWF4基因,其成熟植株花序高度分别增加了35%和14%,与野生型相比,在拟南芥中超表达DWF4基因将增加近两倍的分枝数和结荚数,结子量升高[2]. 而在本实验中,通过在芥菜中超表达DWF4基因,转基因芥菜所结种子数是野生型的近2.5倍,说明超表达DWF4基因可以大幅度促进植株产量提高. 在拟南芥中超表达DWF4基因将使植株叶片叶柄伸长,开花时间提前,转基因植株生长更加旺盛[15]. 在本文图 4和图 5中可以看出,芥菜超表达DWF4基因使植株株高、开展度、最大叶长和最大叶宽皆比同时期野生型增高,转基因植株抽薹开花时间提前. 说明DWF4基因对植株生长发育有促进作用,因此增强了植株生长势,植株生长更加旺盛可能也与转基因植株产量提高息息相关.
-
DWF1诱导24-亚甲基胆甾醇(24-methylenecholesterol)转变为芸薹甾醇(Campesterol)[16],是BR前期合成的重要基因. 在本文中,超表达DWF4基因并没有太多影响DWF1基因的表达,可能是DWF1作用与DWF4基因上游有关. DET2,5-α还原酶被认为是油菜素内酯生物合成酶关键元件,其催化菜油甾醇转化为菜油甾烷醇,是油菜素内酯合成过程中催化较早反应的酶[17]. 在本实验中,超表达DWF4基因促进了DET2的表达,特别是D2和D3植株,表达量超出了野生型植株约2倍,说明DWF4基因正调控DET2的表达. ROT3编码细胞色素P450(CYP90C1),在叶和花的形成发展期间,与细胞极性伸长相关[18],在BR生物合成途径中,ROT3与CYP90D1起着共同催化C23羟基化的作用[19]. 在本实验中,ROT3表达量在转基因植株中表达较高,远远超过野生型植株,说明超量表达DWF4基因促进了ROT3基因的表达. 有报道称CPD基因编码一种重要的BR合成酶(CYP90A1),催化C23羟化反应,代谢油菜甾醇(campesterol)和其他中间产物,Ohnishi等[20]则发现CPD基因参与C3氧化反应. 在本实验中,超表达DWF4基因使得CPD表达量降低,说明DWF4负调节CPD基因,这可能是DWF4基因和CPD基因具有相似性,在BR合成途径中作用位点有相同之处,因此两者产生了抑制作用. CYP85A2调节内源CS水平从而控制BR中C28和C27生物合成途径,在C6氧化途径中,CYP85A2比CYP85A1更加有活性,通过超表达和对两个基因突变体的观察研究也证实了此结论. 在拟南芥中,CYP85A1对CYP85A2起到辅助作用促进BR合成[21],在本实验中,超表达DWF4基因促进了CYP85A1和CYP85A2的表达,而且CYP85A2基因表达量升高更多,佐证了CYP85A2基因比CYP85A1基因活性更强、作用更强的结论,而CYP85A2基因是BR生物合成途径中最后一步反应的重要因子,因此CYP85A2基因表达量的升高证实转基因植株体内BR浓度水平的升高. 所以,超表达DWF4基因促进了植株体内BR合成途径中的相关基因,促进了内源BR的生成.
BAK1编码一个由615个氨基酸组成的蛋白,是一个典型的受体激酶,其在植物体内广泛表达,参与调控BR信号[22],在芥菜中超表达DWF4基因促进了BAK1的表达. BAS1编码一种C26羟化酶,催化CS和BL分别生成26-OH-CS和26-OH-BL,从而使BL功能失活,以维持体内活性BL的静态平衡,在内源BL含量高的组织器官中活跃表达[23]. 但是在本实验中,超表达DWF4基因并没有显著提高BAS1的表达量,其中的内在机理还需进一步研究. 核内蛋白BZR1和其同系物BES1是BR信号途径中的两个重要转录因子,被PP2A蛋白介导脱去磷酸化,去磷酸化后的BZR1和BES1在细胞核中调节将近1 000个靶基因,从而调节植物生长. ChIP-qPCR实验显示BZR1和BES1可以结合DWF4基因和BR诱导基因SAUR-AC1[24]. 在本文中,BES1和BZR1两个基因在转基因植株中表达量远远高于野生型植株,说明超表达DWF4基因促进了转基因植株内源BR提高,因此BES1和BZR1基因转录水平升高从而维持植株体内BR平衡. BR结合BRI1感受器激活BIN2,调节植株生长,BIN2对BES1和BZR1这两个转录因子的磷酸化起着重要作用,是BR信号途径中重要的调控因子[25]. BIN2基因的表达应与BES1和BZR1基因表达相一致. 在本文实验结果中,BIN2基因的表达与BZR1基因表达趋势基本一致,在转基因植株中表达量升高,这与理论结果相同. 拟南芥质膜蛋白组学研究发现BR信号激酶BSKs,磷酸化的BSKs可以和BR受体激酶BRI1相互作用[26]. 在图 8中可以得知,超表达DWF4基因促进了BSKs的表达. 综上所述,通过定量PCR[27]实验结果显示,超表达DWF4基因促进了植株内源BR水平的提高,因此维持体内BR平衡的BAK1,BAS1,BES1,BZR1,BIN2,BSKs基因都各有升高.







 DownLoad:
DownLoad: