-
癌症是一大类疾病的总称,其共同点是失去控制的细胞增殖.在众多的癌症疾病中,多形性胶质母细胞瘤(GBM)是一种神经胶质瘤,是最常见且致死率极高的脑部肿瘤,这种脑瘤具有高侵润性,可大范围转移.同时,该肿瘤细胞对放疗不甚敏感,非常容易复发.据文献报道神经胶质瘤的中位生存时间和无进展生存时间分别为14.6个月和6.9个月,5年生存率为9.8% [1].由于癌症是基因水平上的遗传性疾病[2],所以神经胶质瘤的发生应该是一个多因素作用、多基因参与、经过多个阶段才最终形成的极其复杂的生物学现象.因此,本文从基因层面研究该癌症的致病机制.
从国内外研究来看,在关键基因筛选方面,文献[2]利用STRING工具筛选出一些GBM关键基因;文献[3]通过对GBM患者的基因表达数据进行分析,发现BITC是GBM治疗的潜在生物标志物;文献[4]利用距离加权算法筛选出了与卵巢癌相关的基因.然而,从当前国内外的相关研究来看,没有把关键基因的筛选和GBM患者的生存时间联系起来.由于临床上通常使用无进展生存时间(Progression-free survival)或总生存时间(Overall survival)来对肿瘤等疾病的疗效及预后予以考核,所以把生存时间作为筛选的因素之一.本文拟使用能处理生存时间的生存分析模型[5]来选取和GBM生存时间相关的关键基因,提高基因筛选的准确性.但是由于基因表达数据的高维低样本特点(p≫n),传统的生存分析方法很难对其予以有效的分析,因此我们将以此问题作为起点,对GBM患者的关键基因筛选进行研究.
全文HTML
-
基因表达数据反映的是直接或间接测量得到的基因转录产物mRNA在细胞中的丰度(即反应细胞组织中特定的mRNA平均分子数),可以用来分析基因之间的相关性,推理未知功能的基因,诊断患者的疾病状态,在理解肿瘤发病机制,识别致癌基因,诊断与预防癌症等方面有着重要的应用.
基因表达数据的主要形式是矩阵形式,行表示基因,列表示实验样本[6].数据格式如下所示
Xij表示基因i在不同的实验条件或不同细胞组织j下的表达值,经过数据标准化后,基因表达数据可以进行统计学和生物学分析.
-
本文结合限制性优化算法和生存模型构建了一种新的GBM关键基因筛选算法(SSLC算法),在介绍这种新算法之前,先介绍生存模型和几种限制性优化算法.
-
在医学中,对病人治疗效果的考察,不仅要看疗效的好坏,还要看患者生存时间的长短.生存分析(Survival analysis)是肿瘤和其他慢性疾病疗效评价和预后分析的主要方法[7]. Cox比例风险模型是生存分析方法中最为流行的一种,Cox模型用风险函数来反映协变量对生存期的影响.现假定有n个观测,对每个观测i得到观测值(t,δi,Xi),其中,t为生存时间;δi为截尾指示变量,如果观测i为截尾数据,δi=0,如果观测i为非截尾数据,δi=1;Xi=(xi1,xi2,xip)为p维行向量,表示观测i的第p个协变量. Cox比例风险系数的一般形式为
式中,X=(x1,x2,…,xp)T表示p维协变量向量,β=(β1,β2,…,βp)表示回归系数向量,h0(t)为基准风险函数,则第i个个体的风险率为
其中,X,β,h0(t)的含义同公式(1).
-
经典的Cox风险比例模型擅于处理样本数(n)大于变量(p)的情况[8],然而GBM基因表达数据的变量远大于样本数(p≫n).在我们使用的数据中,样本数为227,基因即变量为10 992.经典Cox风险比例模型不能处理p≫n的数据.因此,我们将能处理p≫n数据的限制性优化算法引入到经典的Cox风险比例模型中,用来处理低样本高变量的GBM基因数据.
1) LASSO(The Least Absolute Shrinkage and Selectionator Operator)算法是限制性优化算法中使用较广泛的经典算法[8].该算法通过构造惩罚函数来获得一个精炼的模型,最终确定一些指标的系数为0. LASSO的公式为
满足
式中,xij表示预测变量,yi表示结果变量,β=(β0,β1,…,βp)表示回归系数向量,t为调整参数.
随后Tibshirani提出了CoxLasso算法[8],即把LASSO和Cox风险比例模型结合,用来处理变量p远大于样本数n且和生存时间相关的数据,可以用来处理癌症基因表达数据.
满足
这等价于以下优化问题:
其中λ为控制稀疏性的调整参数. CoxLasso算法可由R程序中“glmnet”包中的glmnet方法实现.
2) 稀疏主成分算法SPCA(sparse PCA)
主成分分析(PCA)是一类有效的特征提取算法,能够进行特征提取和数据降维.但是,由于主成分分析最终给出的主成分是原来的数据变量的线性组合,因此无法解释每一个主成分对应的特征是什么.特别是把主成分分析应用到基因数据处理时,原始变量可能有成千上万个,那么每一个主成分都是成千上万个变量的线性组合,因此难以解释主成分的含义.
稀疏主成分分析算法解决了这个问题[9].通过增加一个惩罚函数,它会把主成分系数变的稀疏[10],把大多数系数都变成0,通过这样一种方式,就可以把主成分中最重要的部分保留下来,适用于基因数据处理,公式如下:
最大化
满足
X为中心化样本矩阵,A为协方差矩阵,S是某正整数.
-
1) 数据清洗
本文使用的数据来源为Repository for Molecular Brain Neoplasia Data (REMBRANDT) (https://caintegrator.nci.nih.gov/rembrandt)提供的GBM基因表达数据(227个样本),芯片型号为Affymetrix U133 plus 2.0,数据集如表 1所示.
原始的GBM微阵列数据(CEL类型文件)由Bioconductor项目(http://www.bioconductor.org/)提供的R程序(“affy”包中的justRMA方法)进行归一化处理和预处理.经过预处理后,在数据矩阵中有227个样本和54 675个基因;然后,用“GeneFilter”包中的nsfilter方法来筛选出基因表达变化大的基因;最后,GBM的基因表达数据矩阵变为227个样本和10 992个基因.
2) SSLC算法
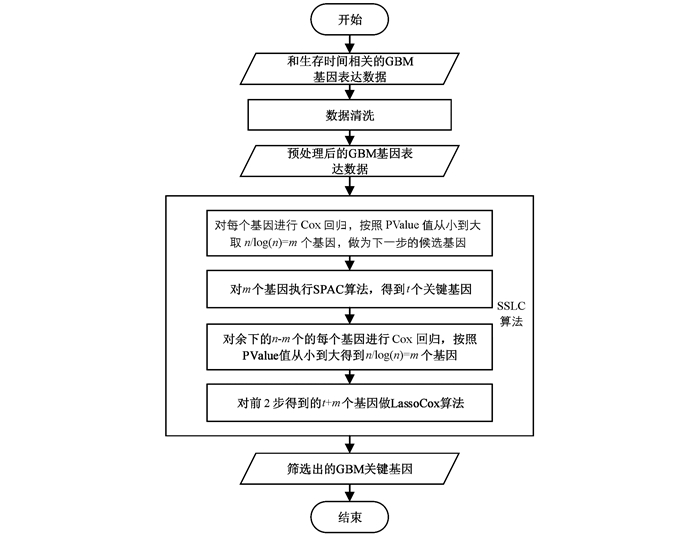
Tibshirani的CoxLasso算法可以用来处理和生存时间相关的高维低样本的GBM基因表达数据,但由实验结果(表 2)可知,由于CoxLasso算法强制把某些参数设置为0,最后筛选出的基因为12个.在未被选中的基因中很可能还存在与GBM相关的基因,这就是假阴性问题.因此我们设计了SSLC算法,通过SIS+SPCA+CoxLasso算法的组合来得到更好的基因筛选结果.本算法由R程序实现.算法流程图如图 1所示,算法步骤如下:
第一,对单个基因(n为样本数,本例n=10 992个)逐个做Cox回归,每个基因得到一个对应的PValue值,对其从小到大排序,然后按照PValue值从小到大取n/log(n)=m个基因(本例m=83),作为下一步的候选基因;选取n/log(n)个基因是为了把p变成比n小,但又不能小太多.实现的关键函数为“glmnet”包中的coxph函数;
第二,对这m个基因执行SPAC算法(稀疏主成分算法),得到t个(本例t=30) 关键基因;实现的关键函数为“nsprcomp”包中的nsprcomp函数;
第三,对余下的n-m(本例10 992-83=10 909) 个基因继续执行步骤一,仍然按照PValue值从小到大得到n/log(n)=m(本例m=83) 个基因;关键函数为“glmnet”包中的coxph函数;
第四,对第二步和第三步得到的t+m(本例30+83=113) 个基因做CoxLasso算法,得到28个关键基因(表 2).关键函数为“glmnet”包中的cv.glmnet函数.
结果如表 2所示:
在SSLC算法(SIS+SPCA+LASSO+Cox算法)中引入了SPCA算法,并反复从候选基因中进行筛选(第一步和第二步,相当于SIS过程),从某种程度上解决了传统的CoxLasso算法假阴性的问题,能为生物医学工作者提供更多的GBM候选关键基因.
-
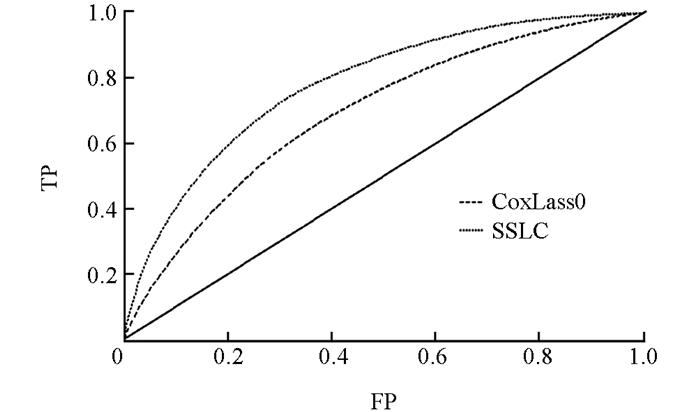
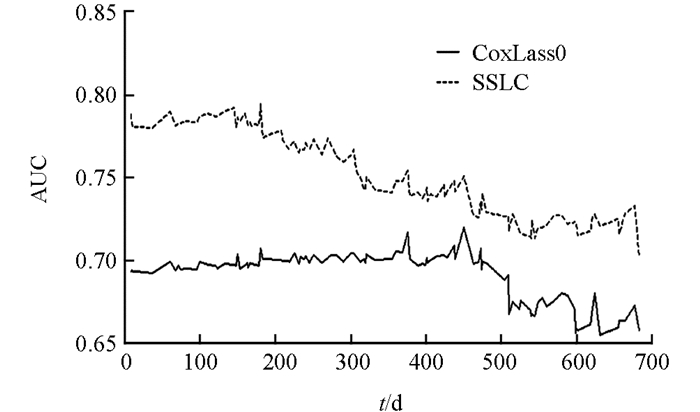
我们利用受试者工作特征曲线ROC(receiver operating characteristic curve)及曲线下的面积AUC来量化2个算法对GBM生存时间预测的准确性[11].
ROC曲线和AUC常被用来评价二值分类器的优劣.一般来说,在ROC曲线中最靠近坐标轴左上方的曲线所代表的分类器准确率最高,在AUC曲线中,曲线下面积越大,诊断准确性越高. ROC和AUC曲线可由R程序的“risksetROC”程序包实现.由图 2、图 3可知,SSLC算法的ROC曲线更靠近坐标轴左上方,AUC曲线下面积也更大,因此SSLC算法要优于传统的CoxLasso算法.
-
在筛选出的关键基因中,查找文献发现,EIF3A在一些癌症中是起正向调节作用的,而且已经发现在一些胶质瘤患者中EIF3A呈高表达[12];IL17RC是一个关键的白细胞介素信号分子,它在GBM的发病过程中对免疫反应和炎症反应起着重要作用[13];GDNF是一种胶质细胞源性神经营养因子,促进神经元存活[14];AEBP1是转录阻遏因子,它在不同组织和器官中的表达水平不同,在脑组织细胞中表现的较强[15],而且它可以与肿瘤抑制蛋白PTEN结合,并抑制其肿瘤抑制功能[16].众所周知,和AEBP1密切相关的PTEN和NF-κB,在GBM肿瘤的进展中起着非常重要的作用.
由此可见,SSLC算法筛选出的基因中有很多是已经证实和GBM高度相关的,还有部分基因虽然现在还未见和GBM相关的文献报道,但可以以此基因为依据选出一些感兴趣的基因作为实验研究的对象.
2.1. Cox风险比例模型
2.2. 限制性优化算法
2.3. GBM关键基因筛选算法——SSLC算法
2.4. SSLC算法和传统的CoxLasso算法的预测性能比较
2.5. 对SSLC算法筛选出的部分基因通过文献查找进行分析
-
现在国内外对GBM关键基因的筛选研究没有和生存时间联系起来,为了解决这个问题,本文把限制性优化算法引入到生存模型中,构建了一种新的GBM关键基因筛选算法(SSLC算法),筛选出了与生存时间相关的GBM关键基因,并比较了SSLC算法与传统CoxLasso算法的区别.最后通过文献查找证明筛选出的部分基因是已经证实的和GBM高度相关的基因.进一步的研究将致力于以这些关键基因为基础,推导出GBM的关键蛋白质,为GBM的靶向制药打下基础.



 下载:
下载: