-
信号传导是基本的生物途径,可将外源信息的变化转化为细胞体内功能的改变[1]. 它控制着包括神经介质和荷尔蒙的释放、调节膜离子通道的开启与关闭、基因转录等重要的细胞活动[2]. 由Gα、Gβ以及Gγ这3条不同链构成的G蛋白控制着生物体内重要的信号传导[1]. 当外源信息发生变化时,与GDP结合的G蛋白分解为Gβγ以及与GTP结合的Gα(GTP-Gα),具有传导活性的Gβγ以及GTP-Gα可实现信号传导,进而实现细胞体内功能的转变[3]. 具有传导活性的物质失活较慢,而RGS蛋白可大大加速这个过程:RGS蛋白与GTP-Gα相结合,加速GTP水解为GDP,进而使得Gα与Gβγ相结合形成没有信号传导活性的与GDP结合的G蛋白[3].
过快的失去信号传导活性会导致一系列的疾病,包括中枢神经系统紊乱、心血管疾病以及癌症[4]. 因此,阻止RGS蛋白与Gα相结合成为药物研发的一个重要方向. 药物可直接作用于RGS蛋白与Gα的PPI界面(protein-protein interface),但是该PPI界面过于平坦以至于并没有可供药物结合的位点[5]. 药物也可以作用于远离PPI界面的RGS蛋白中的氨基酸,通过变构调节实现阻止RGS蛋白与Gα相结合. TDZD抑制剂可与远离PPI界面的半胱氨酸残基通过共价键相结合,通过变构调节来抑制RGS蛋白与Gα相结合[6].
最近的研究表明TDZD抑制剂对RGS蛋白具有一定的选择性,而这种选择性与RGS蛋白中的半胱氨酸残基的数目有关[7]. 然而,研究也表明TDZD抑制剂对具有相同数目半胱氨酸残基的RGS蛋白也有不同的抑制作用. 尽管TDZD抑制剂具有选择性,但选择性较低,进而可能导致药物产生副作用[7]. 因此,需要进一步了解RGS蛋白的氨基酸残基动力学,深入理解RGS蛋白中的药物低选择性以及药理相似性. 本研究使用分子动力学模拟方法,集中深入分析了3种不同RGS蛋白(RGS4,RGS8,RGS17)的结构动力学,在一定程度上解释了药物选择性以及药理相似性的原因.
全文HTML
-
在本研究中,我们使用了VMD (Visual Molecular Dynamics)[8]来建立模型以及分析数据等,NAMD (Nanoscale Molecular Dynamics)[9]来进行分子动力学模拟并使用了CHARMM力场[10]. RGS蛋白的初始坐标来源于PDB (protein data bank):RGS4 (PDB code:1EZT)、RGS8 (PDB code:2IHD)以及RGS17 (PDB code:1ZV4). 我们使用TIP3P水分子对体系溶剂化以及NaCl进行体系的电荷中和. 最终每个体系的原子数目约为30 000.
模拟主要分为3个阶段:首先是500步的体系能量最小化;其次为使用NPT系综进行100 ps的体系体积优化;最后使用NVT系综进行2μs的分子动力学模拟. 在模拟中,时间步长为2 fs,并使用了周期性条件. 通过使用朗之万恒温器,温度控制在310 K;使用Nosé-Hoover恒压器,压力控制在1个标准大气压.
-
为了考察氨基酸残基的柔性,我们计算了均方根涨落(RMSF:root mean squared fluctuation). 较大的均方根涨落表明该氨基酸残基具有较大的柔性,例如处于蛋白质端基的氨基酸,一般来说具有较高的亲水性. 当药物作用于多肽或蛋白质时,也会使得某些氨基酸残基的柔性增大,这表明该药物对这些氨基酸残基的影响较大. 柔性增加的程度越大,表明了该药物对作用的蛋白质影响越大. 我们使用Carma进行了氨基酸残基对的动态互相关(DCC:Dynamic cross correlation)分析[11]. 氨基酸残基对IJ的互相关值(CIJ)定义如下:
$ \mathit{\Delta }{\mathop {{r}_{I}}\limits^ \rightharpoonup}\left( t \right) = {\mathop {{r}_{I}}\limits^ \rightharpoonup}\left( t \right) - \left\langle {{\mathop {{r}_{I}}\limits^ \rightharpoonup}\left( t \right)} \right\rangle $ ,$ {\mathop {{r}_{I}}\limits^ \rightharpoonup}\left( t \right)$ 是氨基酸残基I的α碳坐标,$ \left\langle {{\mathop {{r}_{I}}\limits^ \rightharpoonup}\left( t \right)} \right\rangle $ 为氨基酸残基I的α碳平均坐标. 当2个残基对有75%以上的模拟时间处在4.5Å距离以内时,会形成一个edge (用一条线段表示). DCC分析是变构调节常用的分析方法. 当氨基酸残基对有较高的DCC值时,表明该氨基酸残基对具有较强的(远程)相关作用.我们进一步使用了VMD中的NetworkView插件进行了社区网络分析,在DCC分析的基础上将RGS蛋白划分成了多个社区[12]. 划分方法采用的是Girvan-Newman算法[13]. 最优的社区划分在最大的模块化值取得. 一般而言,模块化值在0.4~0.7之间,本文的模块化值约为0.6.
1.1. 模型以及模拟方法
1.2. 分析方法
-
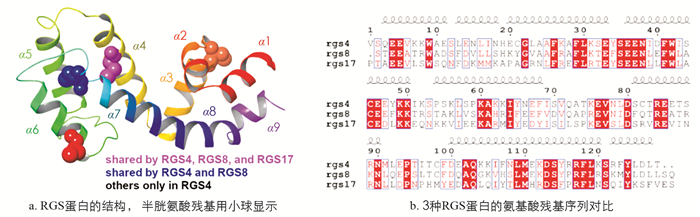
RGS蛋白家族结构上非常相似,具有一个同源的RGS盒子. 该盒子通常含有约120个氨基酸残基,包含了9个α螺旋结构(α1~α9),如图 1所示. RGS4与RGS8在氨基酸序列上有56.3%的相似性,而RGS4,RGS8与RGS17的相似性分别为39.1%以及38.8%(图 1b). RGS4与RGS8高度相似是因为它们同属于R4亚家族,而RGS17属于RZ亚家族[1]. RGS4有4个半胱氨酸残基可供TDZD抑制剂作用,而RGS8具有2个半胱氨酸残基以及RGS17只有1个半胱氨酸残基(图 1a). 3个RGS蛋白具有一个相同位置的半胱氨酸残基,位于α4螺旋上. RGS4和RGS8还具有另一个相同位置的半胱氨酸残基,位于α6~α7之间的无规卷曲(图 1a). RGS4的另外2个半胱氨酸残基分别位于α3螺旋和α6螺旋上.
-
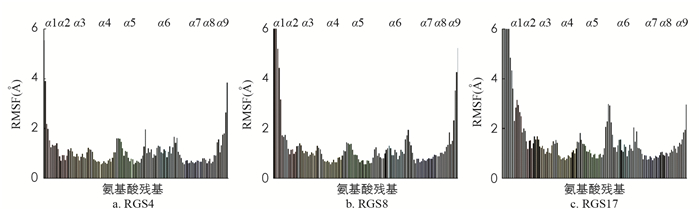
图 2描述了3种RGS蛋白的氨基酸残基RMSF值. 一个大的RMSF数值表明该残基具有强的柔性. 图 2的结果表明端基上的氨基酸残基具有较高的RMSF值,这是由于与端基上的氨基酸残基相互作用的氨基酸较少以及所受到的其他氨基酸的牵制较弱. 同样,我们也可以发现无规卷曲上的氨基酸残基也会有相对较高的RMSF值.
RGS蛋白中与Gα链直接作用的氨基酸残基主要位于α3~α4之间以及α5~α6之间的无规卷曲. 因此,该部分氨基酸残基若具有较大的RMSF值会导致与Gα链较弱地结合,进而使得RGS蛋白与Gα链的相互作用下降,从而表明TDZD抑制剂具有较高的抑制作用. 在图 2中,我们可以发现RGS17的α5~α6之间的无规卷曲具有明显高于RGS4以及RGS8的RMSF值. 如果TDZD抑制剂可以变构调节该无规卷曲,我们可以预测在TDZD抑制剂只结合3种RGS蛋白共同的半胱氨酸残基时,TDZD抑制剂会对RGS17具有相对较高的抑制作用. 而图 2表明3种RGS蛋白在α3~α4之间的无规卷曲的RMSF值差异较小.
同时我们也注意到有3个半胱氨酸残基位于无规卷曲(图 1a中的蓝色小球)或者靠近无规卷曲(图 1a中的橙色和红色小球). 若该无规卷曲具有较大的RMSF值,会使得半胱氨酸残基容易暴露在外,从而易于与TDZD结合,进一步易于抑制RGS蛋白与Gα链的结合. 这种机制也会导致TDZD抑制剂对不同RGS蛋白产生不同的作用机理.
-
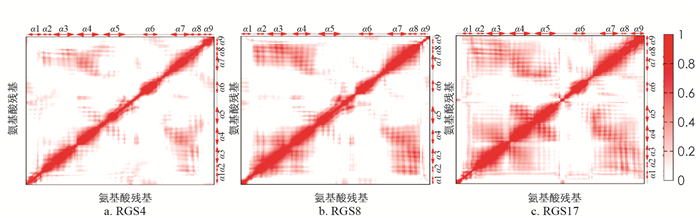
当氨基酸残基对在所有的MD运动轨迹中,运动方向一致时,会给出一个较大的正互相关DCC值,否则给出一个较小的负互相关DCC值. DCC值越大表明该氨基酸残基对相互作用越强,变构调节时二者的相互影响越强. 图 3描述了氨基酸残基对之间的动态正互相关DCC值. 在RGS蛋白中,我们观察到α螺旋之间会具有较大的DCC值,表明该α螺旋对具有较强的(远程)相互影响作用. 当2个靠近的α螺旋具有较大的DCC值时,处于蛋白质内部的残基会较难暴露,进而难以成为药物的作用靶点. 从图 3我们可以看出,3种RGS蛋白的α4螺旋与α7螺旋之间的氨基酸残基对具有较大的DCC值. 这表明位于α4螺旋上的半胱氨酸残基(图 1a中粉色小球)暴露在溶液中的窗口较小,TDZD抑制剂较难作用于该结合位点. 同时我们也发现α4螺旋与α7螺旋之间的氨基酸残基对DCC值有如下关系:RGS17>RGS8>RGS4. 这表明TDZD抑制剂相对更容易作用于RGS4而更难作用于RGS17.
RGS4和RGS8还共同拥有一个位于α6~α7之间的无规卷曲上的半胱氨酸残基(图 1a蓝色小球). 该半胱氨酸残基的暴露涉及α4螺旋与α7螺旋之间以及α4~α5无规卷曲与α6~α7无规卷曲之间的氨基酸残基对DCC值. 从图 3a和图 3b中,我们发现这些DCC值都较小(淡红色)或呈负相关状态(白色). 这表明该半胱氨酸残基容易暴露成为TDZD抑制剂的作用位点.
RGS4中位于α3螺旋始端的半胱氨酸残基也具有较大暴露窗口成为抑制剂的作用位点,这是由于该处与相邻的氨基酸残基对DCC值较小或为负相关(图 3a). RGS4中位于α6螺旋始端的半胱氨酸残基也具有较大暴露窗口. 但该半胱氨酸残基位于RGS4与Gα的PPI界面处,因此该残基较难成为TDZD抑制剂的作用位点.
-
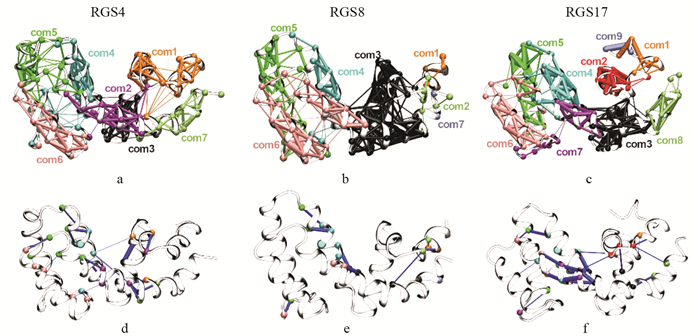
使用Girvan-Newman算法[13]可以将蛋白质划分为多个社区(图 4a,b和c). 一个社区内的氨基酸残基联系紧密且一般运动方向一致(图 4中的粗线段),社区之间的氨基酸残基则关联较弱(图 4中的细线段). 图 4d,e和f描述了各个社区之间的联系.
从图 4中我们发现RGS4和RGS8被划分为7个社区,而RGS17被划分为9个社区. RGS4中α1螺旋与α2螺旋属于同一个社区(图 4a,com1),而RGS8和RGS17的α1螺旋则被划分为多个社区. RGS17的α2螺旋独立为一个社区(图 4c,com2). RGS4中α3螺旋成为一个单独社区(图 4a,com3),而RGS8中α2螺旋、α3螺旋和α8螺旋成立一个社区(图 4b,com3),RGS17中α3螺旋和α8螺旋成立一个社区(图 4c,com3). 这表明RGS8和RGS17中的α3螺旋和α8螺旋联系较为紧密,而RGS4中的α3螺旋和α8螺旋联系较少. 这个现象和DCC图中一致(图 3):RGS8和RGS17的α3螺旋和α8螺旋DCC值较大,而RGS4的α3螺旋和α8螺旋DCC值较小. RGS4和RGS8中α4螺旋没有和α7螺旋中氨基酸残基形成同一个社区(图 4,com4),而RGS17中α4螺旋和α7螺旋中部分氨基酸残基形成了同一个社区. 这个现象也和DCC图中一致. RGS4和RGS8中α4螺旋和α5螺旋中部分氨基酸残基形成同一个社区. RGS8和RGS17中的α5螺旋独立成为了一个社区,而RGS4中的α5螺旋部分氨基酸残基与α6~α7无规卷曲形成了一个社区(图 4,com5). RGS4和RGS17中的α6螺旋独立成为一个社区,而RGS8中α6螺旋和α7螺旋成为一个社区(图 4,com6). RGS4和RGS17中的α7螺旋部分氨基酸残基成为一个社区(图 4a,com2;4c,com7). RGS4和RGS17中的α9螺旋独立成为一个社区,而RGS8中α9螺旋部分残基会和α1螺旋部分残基成为一个社区(图 4a,com7;4b,com2;4c,com8).
从上面分析可以看出,不同RGS蛋白的社区网络存在着相似性,如RGS4和RGS8中(图 4a,4b,com4)、RGS4和RGS17中的(图 4a,4c,com6)和(图 4a,com7;4c,com8)以及RGS8和RGS17中的(图 4b,4c,com3)和(图 4b,4c,com5). 同样不同RGS蛋白的社区网络存在着不同之处,如RGS4的com1和com3、RGS8的com6以及RGS17的com4.
2.1. 结构对比
2.2. 均方根涨落RMSF
2.3. 氨基酸残基对动态互相关DCC
2.4. 社区网络分析
-
药物广谱作用于一个蛋白质家族是药物研发的一个重要方面,提高药物的选择性可以减少副作用的发生[14]. 在本研究中,我们使用了3种RGS蛋白为模型,从氨基酸序列、均方根涨落RMSF、氨基酸残基对互相关DCC以及社区网络分析4个方面给出了3种RGS蛋白的类似性和不同之处,这对于设计新的具有高选择性的药物提供了一定的理论基础,具有一定的指导意义. 本文主要集中讨论了半胱氨酸作为常用的作用位点在不同的RGS蛋白中表现出的相同与不同之处. 研究新的抑制作用位点可能会提高药物的选择性. 同时,本文中的研究方法还可适用于其他的蛋白质家族,如蛋白激酶.
由于有限的计算资源,虽然总的计算时长达到了6 μs,但本文中的蛋白仅限于RGS蛋白,未能考虑RGS蛋白和Gα的相互作用,这是本研究的局限之处. 在未来有足够计算资源的情况下,应对更大的体系进行更长时间的分子动力学模拟[15].



 下载:
下载: