-
开放科学(资源服务)标识码(OSID):

-
奇异变形杆菌是一种广泛存在于自然界的食源性条件致病菌,属于肠杆菌科(Enterobacteriaceae)变形杆菌属(Proteus),无荚膜,无芽孢,革兰氏染色阴性且具有周期性群集运动现象[1]。该菌是一种人畜共患病原菌,具有广泛的宿主嗜性,主要导致人和动物腹泻、食物中毒、胸膜炎、脑膜炎、尿道炎、脊髓炎等病症,其中奇异变形杆菌可导致牛出现以腹泻为主症的临床症状,并引起死亡[2]。近年来,奇异变形杆菌感染动物的相关报道逐年递增,给我国畜牧养殖业带来了不可估量的损失。抗生素类药物滥用及基因组的多样性导致耐药基因和毒力基因频繁转移,为该病的防治带来了巨大挑战[3]。
本研究对前期分离到的5株奇异变形杆菌进行了生物学鉴定、PCR验证、遗传进化同源分析、药敏试验和动物试验,旨在为该菌的病原学研究及引起疾病的免疫防治提供理论依据。
HTML
-
本课题组前期分离出的5株奇异变形杆菌(SPL569、SPL1502、SPL1504、SPL1511、SPL1512),其中SPL569分离自重庆忠县采集的牛鼻拭子,SPL1502与SPL1504分离自四川泸州的牛肺脏组织,SPL1511和SPL1512分离自重庆垫江的牛肛门拭子。
-
细菌DNA提取试剂盒,北京天根生化科技有限公司;普通琼脂培养基、马丁肉汤琼脂培养基,海博生物(青岛)公司;肠杆菌生化鉴定管、药物敏感纸片,杭州微生物公司;琼脂凝胶电泳仪,Bio-rad公司;蛋白成像系统,Tanon公司;智城ZWY-2102C恒温摇床,上海智城分析仪器制造有限公司;隔水式恒温培养箱,上海齐欣科学仪器有限公司;雌性18~22 g昆明小鼠(4~6周龄),恩斯维尔公司。本研究涉及的引物均由生工生物工程(上海)有限公司合成,所有试验操作均在P2实验室进行,动物试验加伦理审核编号为IACUC-20221114-08。
-
从-80 ℃超低温冰箱中取出冻存菌株,划线于马丁固体培养基上,37 ℃恒温培养箱培养18~20 h。待单菌落长出后,挑取单菌落于5 mL马丁液体培养基中,37 ℃恒温摇床220 r/min培养8 h,置于-4 ℃冰箱中备用。
-
取马丁固体培养基上的纯培养物分别接种于肠杆菌科细菌微量生化鉴定管中,37 ℃恒温培养箱培养24~28 h,观察并记录试验结果。
-
取培养好的菌液2 mL,使用细菌DNA提取试剂盒提取总DNA。查阅GenBank中ureR(AM942759)基因序号,应用BioXM 2.6软件设计奇异变形杆菌特异性引物并对其进行PCR扩增,引物序列见表 1。
-
用细菌16S rDNA的通用引物进行PCR扩增,引物序列见表 2。反应总体系(50 μL):2×TaqMix 24 μL,上、下游引物各2 μL,DNA模板2 μL,ddH2O 20 μL。反应条件:95 ℃预变性3 min;95 ℃变性30 s,57 ℃退火30 s,72 ℃延伸90 s,共35个循环;72 ℃延伸10 min。扩增产物经1.0%琼脂糖凝胶电泳检测,胶回收产物送生工生物工程(上海)有限公司测序。
-
将所测试验菌株的16S rDNA序列与NCBI数据库中收录的序列进行比对和同源性分析,并运用DNAStar和MegaX软件构建系统发育树。
-
群集运动距离和速度参照Liaw等[4]介绍的方法进行,比较试验菌株和奇异变形杆菌属性的相符性。为了更好地观察群集运动的轨迹及细胞呼吸状态,本研究往不同浓度的琼脂培养基中添加了终浓度为0.005%的TTC(2,3,5-氯化三苯基四氮唑);同时挑取显色区和未显色区的菌苔进行革兰氏染色,观察二者菌体间的差别。
-
取30只昆明小鼠,随机分为6组,设试验组(5只)和对照组(5只),试验组将试验菌SPL569、SPL1502、SPL1504、SPL1511、SPL1512菌液培养物按0.1 mL/只(细菌浓度分别为1.67×109 CFU、1.48×109 CFU、2.36×109 CFU、1.54×109 CFU、1.34×109 CFU)剂量腹腔注射小鼠,对照组腹腔注射无菌液体培养基。各组接种后隔离饲养,自由饮水进食,每隔6 h观察1次小鼠的临床表现,连续观察1周。对各组出现症状及死亡的小鼠进行剖检观察,分离鉴定病变脏器内的细菌。
-
基于5株奇异变形杆菌的致病性存在差异,为探究致病性与毒力基因间的关系,通过查阅文献设计了PM编码尿素酶(ureC)、MRP菌毛(mrpA)、金属蛋白酶(zapA)、适温菌毛合成分子(atfC)、适温菌毛(atfA)、Pmf菌毛(pmfA)、尿道上皮粘附素(ucaA)、“雾蔓”迁徙能力调节因子(rsbA)等8种奇异变形杆菌潜在的毒力基因特异性引物(表 3),以5株试验菌的DNA为模板,对其毒力基因进行PCR扩增。扩增总体系(20 μL):2×TaqMix 10 μL,上、下游引物各1 μL,DNA模板1 μL,ddH2O 7 μL,反应参数见表 4。扩增产物经1.0%琼脂糖凝胶电泳检测,并观察结果。
-
采用纸片扩散法(K-B法)在马丁琼脂(含10%马血清)培养基上测定分离病原菌对药物的敏感性。参照杭州微生物试剂公司《药敏试验纸片法的抑菌范围解释标准》,根据抑菌圈的大小将细菌分为敏感(S)、中介(I)和耐药(R)[5]。
-
鉴于SPL569、SPL1502、SPL1504、SPL1511、SPL1512菌株对临床常用抗生素均产生耐药,为了研究细菌耐药表型与耐药基因之间的关系,本研究设计了相关耐药基因的特异性引物(表 5),以5株试验菌的DNA为模板,对其耐药基因进行PCR扩增。PCR扩增体系同毒力基因检测,反应参数见表 6。扩增产物经1.0%琼脂糖凝胶电泳检测,并观察结果。
1.1. 材料
1.1.1. 菌株来源
1.1.2. 主要试剂及试验动物
1.2. 方法
1.2.1. 菌株复苏
1.2.2. 生化试验
1.2.3. PCR特异性引物扩增
1.2.4. 16S rDNA的PCR扩增与测序
1.2.5. 16S rDNA序列同源性分析
1.2.6. 周期性群集运动的检测
1.2.7. 小鼠致病性试验
1.2.8. 菌株毒力基因检测
1.2.9. 药敏试验
1.2.10. 菌株耐药基因检测
-
菌株生化试验结果见表 7。参考《伯杰氏系统细菌学手册》,初步判定5株试验菌均属于奇异变形杆菌科[6]。
-
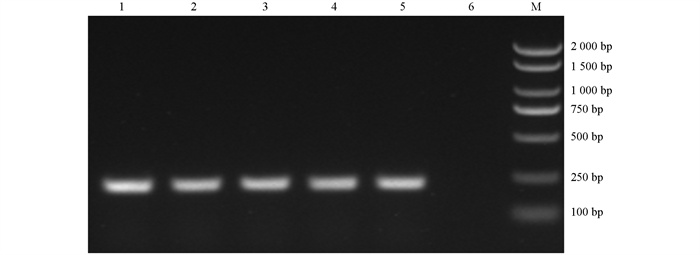
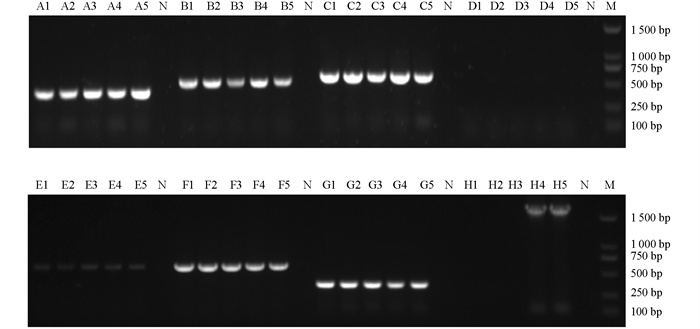
PCR扩增产物经1%凝胶电泳后结果见图 1,条带大小与预期结果相符,结合生化试验结果可以判定5株试验菌均为奇异变形杆菌。
-
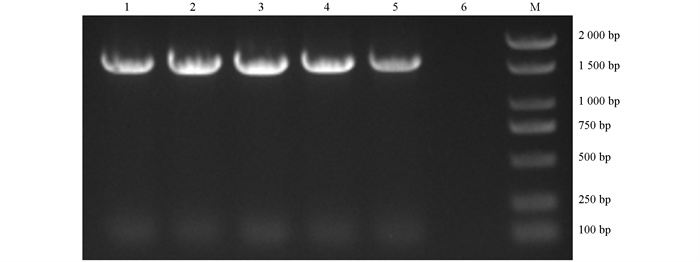
分别以试验菌株SPL569、SPL1502、SPL1504、SPL1511、SPL1512的总DNA为模板,利用细菌16S rDNA通用引物进行PCR扩增,1%琼脂糖凝胶电泳后,得到大小为1 450 bp的扩增片段,结果符合预期大小(图 2)。
将扩增产物测序所得的序列在NCBI上进行Blast比对,下载同源性较高的序列通过Clustral W比对后,使用MegaX进行进化树的构建,建树方法为NJ,选择变形杆菌科5个已经命名的种属与试验菌株进行同源分析。从进化树结果(图 3)可以看出,5株试验菌均与奇异变形杆菌的亲缘关系最近。
-
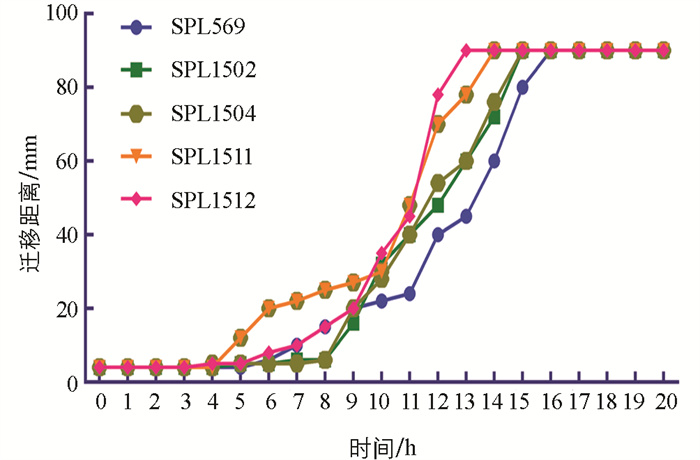
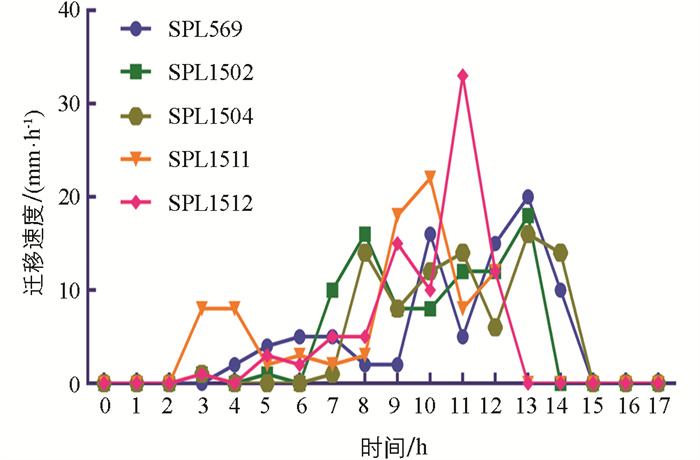
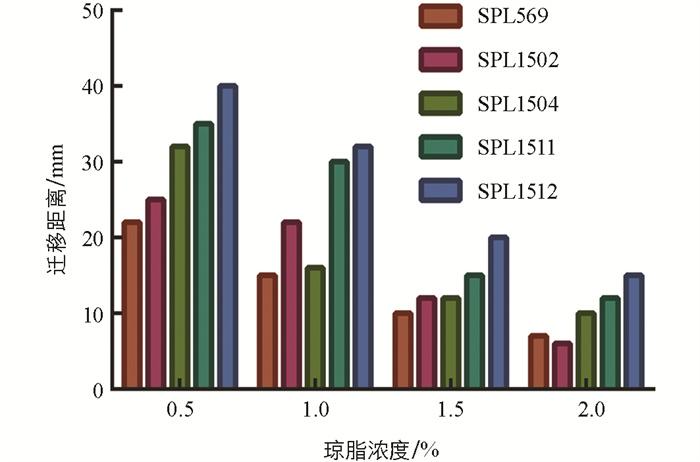
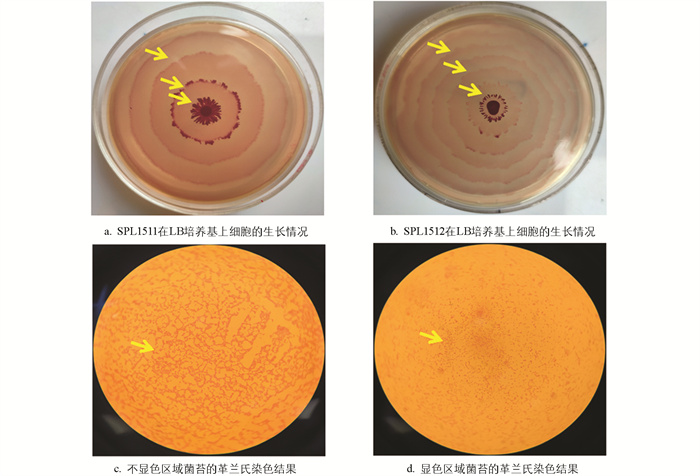
将试验菌株接种于1.5%琼脂LB固体培养基中心,4 h后菌斑由透明变成乳白色并向四周迁移扩散,16 h后在培养基表面形成多圈状扩散同心圆菌苔(图 4)。分离株培养至4 h左右开始发生群集迁移运动,迁移距离随着时间的延长而增大,在14 h时基本达到生长停滞期(图 5)。迁移速度呈锯齿样波动,在8~12 h迁移加快,随后开始下降,最终在15 h后达到最小值0,其中SPL1511和SPL1512迁移速度相对较快,推测可能跟后面检测到的“雾蔓”迁移基因rsbA有关(图 6)。将试验菌株分别接种于含0.5%、1.0%、1.5%、2.0%琼脂的LB固体培养基上37 ℃培养8 h,在0.5%琼脂培养基上迁移距离最远,呈现出琼脂浓度越高,迁移距离越短的现象(图 7)。选取迁移现象明显的SPL1511和SPL1512接种于含TCC指示剂的1.5%琼脂LB固体培养基上,试验菌株形成了红色同心圆扩散现象,清晰地展示了在群集运动过程中呼吸酶活性的周期性变化,红环代表的是呼吸酶活性正常将TCC还原成红色不溶物的现象,不显色区域是呼吸酶被抑制的结果(图 8)。革兰氏染色可以看到,在不显色区域的菌体呈长杆状,显色区域的菌体呈短杆状,与预期相符。
-
试验菌人工腹腔感染试验鼠,每2 h观察1次,记录其临床表现。2 h后试验组SPL1511、SPL1512出现了精神沉郁、毛发杂乱、蜷缩抱团、眼角出现分泌物等临床表现,其他组未见明显症状。菌株SPL1511组最早6 h出现死亡现象,48 h内全部死亡;菌株SPL1512组最早4 h出现死亡现象,24 h后全部死亡;对照组无明显临床症状,全部存活(表 8)。对试验组死亡的小鼠进行无菌病理剖检采集病料,从小鼠的心血、肝脏、肾脏、肺脏中均分离到一致的菌株,且菌落形态、菌体形状、16S r DNA PCR结果以及基础的理化特征与感染的试验菌株一致(图 9);对照组小鼠对应的脏器中未分离到细菌。
-
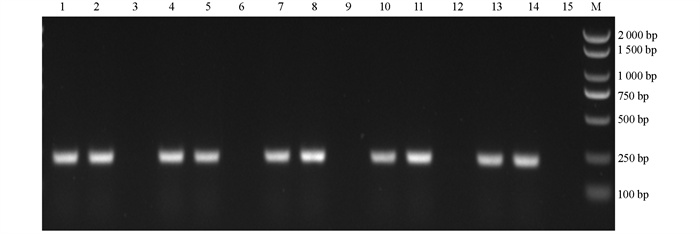
5株试验菌毒力基因的检测结果(图 10)显示,毒力基因atfA、atfC、zapA、pmfA、ureC、ucaA的携带率为100%,未携带毒力基因mrpA,而毒力基因rsbA的阳性携带率为40%,仅LPS1511、LPS1512携带。
-
临床常用的20余种抗菌药物的敏感性试验结果(表 9)显示:5株试验菌对临床常用的大部分抗菌药物均有一定的耐药性,其中SPL569、SPL1502、SPL1504菌株只对头孢曲松敏感,SPL1511只对头孢他定敏感,SPL1512只对头孢哌酮敏感。
-
为探究5株试验菌耐药表型与耐药基因之间的关系,本研究对奇异变形杆菌的8大类24种耐药基因进行验证,分别为β-内酰胺类(CTX-M、TEM、SHV、cmy、bla PSE、bla OXA),磺胺类(sul1、sul2),氨基糖苷类(Aac(6’)-Ib-cr、aadA),喹诺酮类(qnrA、qnrB、qnrS),多粘菌素类(vanA、vanB、vanC),大环内酯类(ermB、mefA、mrsD),四环素类(tetA、tetM),氯霉素类(cmlA、stcM、floR)[7]。分别以5株试验菌的基因组DNA为模板对耐药基因进行扩增(图 11),其中β-内酰胺类TEM、bla PSE、bla OXA基因,磺胺类sul1、sul2基因,氨基糖苷类Aac(6’)-Ib-cr、aadA基因,喹诺酮类qnrA基因,氯霉素类cmlA、stcM、floR基因均被检测到,且TEM、sul1、sul2、Aac(6’)-Ib-cr、aadA、stcM等耐药基因的携带率为100%,耐药基因和耐药表型相符。
2.1. 生化鉴定结果
2.2. 奇异变形杆菌特异性引物PCR扩增结果
2.3. 16S rDNA PCR扩增结果及测序分析
2.4. 群集运动分析
2.5. 小鼠致病性试验结果
2.6. 试验菌株毒力基因检测结果
2.7. 药物敏感性试验结果
2.8. 5株试验菌的耐药基因检测结果
-
变形杆菌属归于肠杆菌科[8],已命名的有5个种,分别为奇异变形杆菌(Proteus mirabilis)、普通变形杆菌(Proteus vugaris)、潘氏变形杆菌(Proteus penneri)、粘化变形杆菌(Proteus myxofaciens)和豪氏变形杆菌(Proteus hauseri),其中又以奇异变形杆菌(Proteus mirabilis)与临床关系尤为密切[9]。该菌主要有内源性和外源性两种感染方式,内源性感染主要是当机体因外界环境变化导致免疫力下降时,体内的奇异变形杆菌大量繁殖而导致发病;外源性感染主要是被污染的水源、食物或空气经消化道、呼吸道等方式进入感染宿主[10]。猪、犬、羊等家畜都是易感动物,关于牛感染奇异变形杆菌的报道也在逐年增加,且多以继发感染为主,给我国养殖业发展带来了阻碍。值得注意的是,该菌同样可以感染人,可导致腹泻、脊髓炎、尿道炎、败血症等疾病[11]。本研究将分离出的5株奇异变形杆菌通过革兰氏染色、生化试验鉴定、特异性引物扩增和16S序列同源分析等方法进行了验证,发现5株试验菌与奇异变形杆菌(Proteus mirabilis)的同源性最高。
奇异变形杆菌的群集迁移运动(Swarming motility,SM)与其致病性及抵抗外界侵袭能力息息相关[12]。当SM发生时,奇异变形杆菌分化成潜生体(Cryptic growth cell,CGC)和繁殖体(Vegetative cell,VC)两种形态[13]。当细胞感受到来自外界环境变化的信号后表面开始诱导分化,此时VC生长活跃产生大量酸性代谢物,环境不利于细菌生长,因此转录活化因子FlhD/FlhC被激活,抑制了细胞呼吸酶的活性,细胞分裂受到抑制;为适应环境变化,VC开始分化成CGC,大量鞭毛蛋白由菌细胞合成并在鞭毛旋转运动信号的控制下向代谢废物浓度较低的外围扩散;待其扩散至代谢物少、营养丰富区域时,FlhD/FlhC由于半衰期短已被水解,呼吸酶活性再次增强,CGC向VC转化,此过程的交替反复出现导致了同心圆菌苔的产生,迁移速度也呈现锯齿样波动[14]。奇异变形杆菌的迁移速度与培养基表面的水分及琼脂的浓度密切相关,当琼脂浓度较低时(0.5%、1.0%),培养基表面水分较多,有利于代谢物扩散,奇异变形杆菌在接种处生长成VC状态,由于CGC亲水性较强,并迅速向外扩散,等CGC状态铺满整个板面时再转化成VC状态;当琼脂浓度较高时(1.5%、2.0%),代谢物不容易扩散,VC和CGC状态交替出现,形成了透明和乳白色同心圆现象。细菌在正常呼吸过程中脱氢酶会产生氢,2,3,5-氯化三苯基四氮唑(简称四唑或TTC)可以在细胞的还原过程中与氢结合发生氢化作用生成一种不溶于水、稳定的、不扩散的红色物质三苯基甲(TTCH)。脱氢酶活性与细胞的呼吸有关,而TTCH的生成量与细菌代谢活力的强弱又呈正相关,因此,可根据红色深浅所反映的脱氢酶活性作为细菌代谢活力的指标[15]。奇异变形杆菌的群集迁移能力减弱会导致侵袭能力减弱,而没有群集迁移能力的菌群甚至会丧失侵袭能力。本试验5株试验菌中SPL1511和SPL1512群集迁移能力比其他3株强,且只有SPL1511和SPL1512对小鼠致死,进一步验证了群集运动能力是影响其侵袭力和致病性的关键。
细菌的毒力因子是宿主致病的重要因素。据报道奇异变形杆菌的毒力因子主要有:菌毛、溶血素、脲酶、免疫逃避、金属摄取等[16]。本研究选取相关的8个毒力基因ureC、zapA、ucaA、rsbA、mrpA、pmfA、atfA、atfc进行验证,最终atfA、atfC、zapA、pmfA、ureC、ucaA均被检测到,MRP菌毛主要结构亚单位mrpA基因未被检测到。有趣的是,只在SPL1511和SPL1512菌株中检测到独特的“雾蔓”迁徙能力调节因子(rsbA)。“雾蔓”迁徙能力是奇异变形杆菌的一种非常重要的毒力特征,rsbA与“雾蔓”迁徙能力密切相关,群集运动试验结果也显示这2株菌的迁移能力强于其他菌株,而群集迁移能力又与侵袭力正相关,再结合致病性试验结果,得出这2株菌具有强致病性,推测rsbA基因与奇异变形杆菌的致病性有关,具体作用机制还有待进一步研究。
群集运动有助于细菌逃避抗菌剂及免疫系统的杀伤作用,使细菌产生耐药性。众所周知,细菌在高细胞密度时可以耐受较高的抗菌剂浓度,但是运动能力及速度赋予了其更大的生存优势[17]。获得性耐药与运动速度存在直接关系,细菌运动速度越快,暴露在抗菌剂中的时间越短,越不容易死亡,耐药性就越强,因此具有群集运动的细菌表现出更高的耐药性。本研究结果表明,5株试验菌均具有群集运动现象,对临床常见的大部分抗生素都表现出耐药性,其中具有更强群集运动能力的SPL1511和SPL1512对比其他3株试验菌对头孢曲松表现出更强的耐药性。对8类24种耐药基因的检测发现,耐药表型跟耐药基因型符合率较高。由此可见,5株试验菌表现出的耐药性与奇异变形杆菌的群集运动存在一定的联系。通过药敏试验表明,耐药基因介导了耐药表型的发生,通过耐药基因与表型的共筛选从而对敏感药物进行预测,可为临床用药提供可靠的理论指导,减少多重耐药菌株的产生。








 DownLoad:
DownLoad: