-
生姜(Zingiber officinale Rosc.)为姜科(Zingiberaceae)多年生宿根性草本植物,是主要香辛保健蔬菜之一,也是医药及化工产品的重要原料[1]. 我国生姜栽培历史悠久,是世界上生姜生产、出口、消费第一大国[2]. 近年来,我国生姜种植面积不断增大,但生姜产区连年重茬,各种病害日益严重. 其中,生姜青枯病又称姜瘟病,是一种世界性的土传细菌病害,严重制约了生姜产业的发展[3].
青枯病是一种严重危害作物种植生产的毁灭性土传细菌病害,其病原菌寄主范围广,可侵染包括生姜、番茄、辣椒、茄子、马铃薯等50个科的400多种植物[4-5]. 生姜青枯病的病原菌形态结构、种内分化和致病机理复杂[6],其发生与流行受土壤湿度、温度和种植方式等多种因素的影响[7],目前尚无理想的防治手段和生姜抗病品种[8]. 前人采用组织分离法已对安徽[9]、四川[10]、海南[11]等地的生姜青枯病病原菌进行过鉴定. 湖北省作为生姜的重要种植地区,近年来青枯病频发,田间发病率在30%~50%,严重时甚至绝收. 然而,关于湖北生姜青枯病病原菌的研究仍鲜有报道. 为此,明确湖北生姜青枯病的病原菌,可为该病有效防治提供基础.
为防止带菌姜种和带菌土壤引起的生姜青枯病的传播,建立快速准确的生姜青枯病菌检测方法对生姜种植过程重大土传病害的早期检测与预警防控有重要意义. 传统的青枯病病原菌检测包括症状观察、分离纯化等手段,操作复杂且时效性差,还极易与其他病害混淆,难以快速准确鉴定,不能满足生产实际的需要[12]. 分子检测方法已成为当前主要的病原菌检测手段,不仅能应用于田间病害的早期诊断预警,而且还能满足种子潜伏期检疫的要求[13]. PCR检测技术是最常用的植物病原菌分子检测方法,具有成本低、灵敏度高、特异性强、稳定性好等优点,已在植物组织及土壤病原菌检测方面得到了广泛运用[14]. 例如,钟鑫等运用普通PCR技术分子体系在抗病品种接种1~5 d后检测出西瓜枯萎病病菌[15],Martínez-Espinoza等利用PCR技术在玉米染病早期或者无症状感染时检测玉米瘤黑粉病病菌[16],张凯东等采用PCR和序列测定技术对柑桔叶样进行了黄龙病菌检测等[17]. 迄今为止,生姜青枯病的PCR检测技术尚未见报道.
为了明确引起湖北恩施地区生姜青枯病的病原菌以及建立快速准确的检测方法,以取自湖北恩施地区的生姜病样进行分离纯化,基于形态学特征、致病性测定和分子生物学的方法对其进行鉴定. 检索查询国内外关于青枯菌的分子检测报道,筛选了青枯雷尔氏菌的特异性扩增引物,优化了PCR快速检测方法,旨在为生姜青枯病害的田间早期预警、姜种检疫诊断及绿色防控提供技术依据.
HTML
-
疑似生姜青枯病病株采自湖北恩施(109°41′48″E,30°03′30″N),健康生姜植株种于长江大学玻璃温室中. 生姜青枯雷尔氏菌(Ralstonia solanacearum)标准菌株由中国农业科学院植物保护研究所提供;番茄青枯菌(R. solanacearum)、土豆青枯菌(R. solanacearum)、藿香青枯菌(R. solanacearum)、烟草青枯菌(R. solanacearum)、变黄假单胞菌(Pseudomonas flavescens)、台湾假单胞菌(Pseudomonas taiwanensis)、谷关假单胞菌(Pseudomonas entomophila)、大黄鱼致病性香鱼假单胞菌(Pseudomonas plecoglossicida)、胡萝卜软腐果胶杆菌(Pectobacterium aroidearum)、路氏肠杆菌(Enterobacter ludwigii)、蜡样芽孢杆菌(Bacillus cereus)、鲍曼不动杆菌(Acinetobacter seifertii)、成团泛菌(Pantoea agglomerans)、解鸟氨酸拉乌尔菌(Raoultella ornithinolytica)均由长江大学香辛作物研究院实验室保存.
-
2019年8月于湖北省恩施土家族苗族自治州宣恩县生姜种植基地采集具有典型青枯病症状的生姜植株,将病样带回实验室进行病原菌分离.
生姜青枯病病原菌的分离参照任欣正等[18]的方法,略有改进. 用无菌手术刀截取生姜病株根茎部的病健交界处组织,剥去外表皮,置于装有50 mL无菌水的三角瓶中,37 ℃、150 r/min振荡,待无菌水稍有浑浊后,用接种环蘸取少量液体,在TTC培养基平板上划线培养,放置于30 ℃恒温培养箱中培养48 h,长出的菌落用于后续的致病力测定.
-
将分离得到的病菌在TTC培养基平板上重新划线,30 ℃培养36 h后挑取单菌落在TTC液体培养基中,30 ℃、200 r/min条件下培养24 h,调整细菌浓度为107 cfu/mL后进行致病力测定试验. 选取生长60 d且长势一致的盆栽生姜苗,用无菌的4号注射器吸取1 mL菌液,采用茎基部注射接种法在生姜植株的茎基部进行接种,以无菌水为阴性对照,每个处理重复接种20盆. 在接种第3 d后调查发病情况,在接种第15 d后计算病情指数. 青枯病病情共分成5个等级[19]. 0级:植株长势健康;1级:1个叶片枯萎;2级:2至3个叶片枯萎;3级:3片(含)以上叶片枯萎;4级:植株死亡. 统计病株率及病情指数,判定致病力强弱. 根据柯赫氏法则重新对接种发病的生姜植株进行病原分离鉴定,观察与原分离病菌是否一致. 选取致病力强的菌株进行病原菌的下一步鉴定.
-
形态学鉴定:参照《常见细菌系统鉴定手册》[20],将平板上菌落颜色和形状与青枯雷尔氏菌相似的单菌落重新接到TTC平板上,观察菌落的形态特征,并进行革兰氏染色,在光学显微和扫描电镜下观测其形态特征.
分子生物学鉴定:采用CTAB/NaCl法提取菌株基因组DNA,以提取的DNA为模板,通过细菌16SrDNA通用引物16S-F/16S-R(16S-F:5'-AGAGTTTGATCCTGGCTCAG-3',16S-R:5'-TCGGCTACCTTGTTACGACAC-3',由武汉华大基因科技服务有限公司合成)进行PCR扩增,标准菌株为阳性对照,无菌去离子水为阴性对照. PCR扩增程序为:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,72 ℃补齐10 min,循环30次. 扩增后的PCR产物经琼脂糖凝胶电泳纯化回收后,送武汉华大基因科技服务有限公司进行双向测序,并对测序的结果进行拼接,得到菌株的16S rDNA扩增片段的序列信息,并将其在NCBI上进行BLAST比对分析. 另外,在NCBI上选取不同细菌的16S rDNA序列,使用MEGA7.0软件,通过邻接法(neighbor-joining method)来构建分离菌株的系统发育树.
生理小种和生物型鉴定:根据菌株对不同寄主的致病性确定所属的生理小种[21]. 选取生长10 d的生姜、番茄、茄子、马铃薯、烟草和辣椒的健康植株,采用茎基部接种法,在植株茎基部注射浓度为107 cfu/mL的菌液,以注射无菌水为阴性对照,每种植物重复接种10盆. 在接种后的第7 d记录发病植株数量,并以发病率为指标来统计青枯菌对不同植物的致病力. 参考吕志强等[22]的青枯雷尔氏菌生物型划分标准,分别以乳糖、麦芽糖、纤维二糖、甘露醇、山梨醇和甜醇作为培养基的碳源,根据菌株对碳源的利用情况来确定生物型.
-
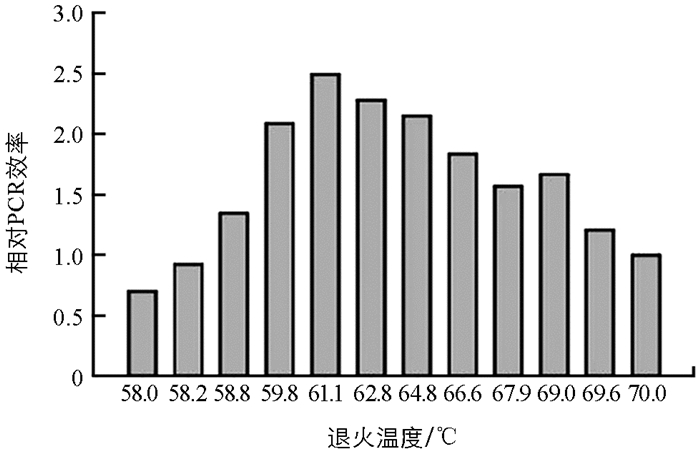
根据国内外关于生姜青枯病病原菌的分子检测报道,筛选出特异性引物21R/21F(21F:5'-CGACGCTGACGAAGGGACTC-3';21R:5'-CTGACACGGCAAGCGCTCA-3')[23],引物由武汉华大基因科技服务有限公司合成. 对影响PCR方法的退火温度、延伸时间及循环次数重要因素进行优化. 选用不同的退火温度58.8 ℃设计PCR反应程序,共设计58 ℃,58.2 ℃,58.8 ℃,59.8 ℃,61.1 ℃,62.8 ℃,64.8 ℃,66.6 ℃,67.9 ℃,69.0 ℃,69.6 ℃,70 ℃ 12个温度梯度,3个延伸时间分别设为30 s,45 s和1 min,3个循环次数分别设为30,35,40次,每个因素设置3个重复. 结果按照病原菌扩增的特异性和敏感性,即条带的强弱、杂带的有无,并结合Alphalmaher TM 2200软件进行综合评价[24].
-
特异性检测:对分离得到的生姜青枯菌基因组DNA,分别以番茄青枯菌、土豆青枯菌、藿香青枯菌等12株菌株基因组DNA为对照,利用优化好的PCR反应方法进行扩增. 根据电泳条带的有无检测生姜青枯菌引物的特异性,并且对阳性样品的扩增产物进行双向测序,测序结果进行BLAST比对分析. 试验设置3次重复.
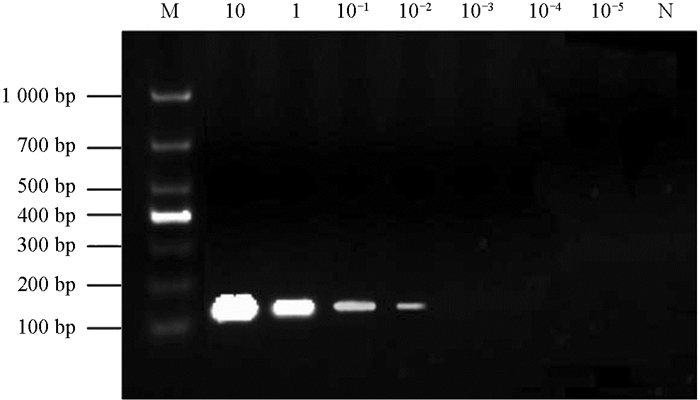
灵敏度检测:利用超微量紫外分光光度计测定青枯菌模版DNA纯度及浓度,将模板浓度稀释至10,1,10-1,10-2,10-3,10-4,10-5和10-6 ng/μL共8个浓度梯度,使用优化后的PCR方法进行PCR扩增,取5 μL扩增产物进行琼脂糖凝胶电泳,凝胶成像系统照相. 试验设置3次重复.
-
田间采集具有典型生姜青枯病症状的病株及其根际土壤样本(表 1),提取生姜病株组织和土壤基因组DNA后用建立的PCR方法进行病原菌检测. 同时,对采集的样本进行病原菌分离培养鉴定,以验证该PCR检测方法的准确性. 试验设置3个重复.
-
采用SPSS软件对试验数据进行分析(p < 0.05),绘图采用sigma plot软件.
1.1. 材料
1.2. 方法
1.2.1. 生姜病样采集与病原菌分离
1.2.2. 致病力测定
1.2.3. 生姜青枯病病原菌鉴定
1.2.4. 生姜青枯病病原菌PCR检测方法的建立及优化
1.2.5. PCR反应特异性和灵敏度检测
1.2.6. 植株组织和土壤的病原菌检测
1.3. 数据分析
-
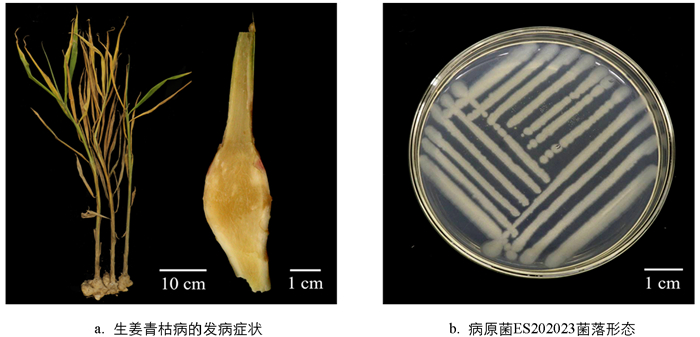
从采集具有典型青枯病症状的生姜植株(图 1a)中分离得到了10株培养性状一致的菌株,菌落为圆形或梭形,菌体乳白色,表面光滑,略有隆起(图 1b). 选取3个典型菌株ES202021,ES202022和ES202023作为代表菌株进行后续致病力测定.
-
以活化1 d的ES202021,ES202022和ES202023菌悬液接种到培养60 d的健康生姜苗,以接种无菌水为空白对照,观察并记录生姜植株的发病情况. 结果显示,接种菌株后生姜叶片边缘开始内卷下垂、叶尖变成黄色,然后扩展至整个叶片,导致叶片逐渐枯萎,最后整个植株死亡,这与所采集的生姜发病植株的发病症状表现一致,而对照植株完全不发病(图 2). 15 d后调查病情指数,结果显示,接种菌株ES202021,ES202022和ES202023的生姜苗病情指数分别为35.2,34.1和38.2,平均病情指数超过30.0. 3个菌株均具有强致病力,其中ES202023致病力最强,选取其为代表菌株进行下一步的鉴定. 分别收集接种菌株ES202023和无菌水培养5 d后的生姜植株组织,进行重新分离纯化鉴定,其试验结果表明,在接种菌株的生姜植株组织中分离纯化获得的病菌与接种菌株ES202023相同,而接种无菌水的空白对照组织样本中没有分离到接种病原菌,符合柯赫氏致病性验证法则.
-
ES202023在培养基上单菌落为圆形或梭形,菌体乳白色,表面湿润有光泽,流动性极强,生长到第3 d时,菌落中间出现粉红色. 革兰氏染色结果显示,该菌株为革兰氏阴性菌(图 3a). 扫描电镜结果显示,菌体为单胞,呈短杆状,大小为(0.5~0.7) μm× (1.5~2.0) μm(图 3b).
-
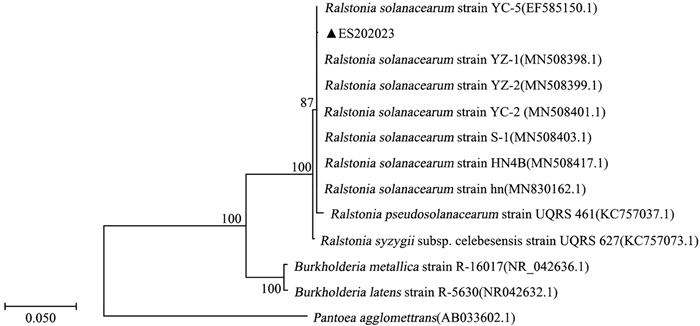
以提取的病原菌基因组为模板,通过通用引物16S-F/16S-R进行PCR扩增其16SrDNA序列,得到约1 400 bp的特异扩增片段. 将片段测序结果在NCBI数据库进行BLAST对比,如表 2所示,ES202023与已经报道的青枯雷尔氏菌序列相似性为99.9%. 下载相似度较高的多条序列、同属序列和外群序列的16SrDNA序列,构建菌株ES202023的系统进化树(图 4). 结果表明,菌株ES202023与多条青枯雷尔氏菌聚类在同一分支上. 结合形态学特征、致病性测定与分子生物学鉴定,证明菌株ES202023为青枯雷尔氏菌(R. solanacearum).
-
菌株对不同寄主的致病性结果显示,分离获得的青枯雷尔氏菌ES202023对生姜和番茄具有强致病力,发病率为100%,对马铃薯具有弱致病力,发病率30%,不能感染茄子、烟草和辣椒. 根据Hayward[25]的生理小种划分标准,ES202023菌株属于4号生理小种. 菌株对碳源的利用结果显示,分离获得的青枯雷尔氏菌ES202023对麦芽糖、纤维二糖、乳糖、甘露醇、山梨醇和甜醇6种碳源的利用结果均呈阳性反应,依据Hayward[25]的生物型划分标准,ES202023菌株属于生物型Ⅲ.
-
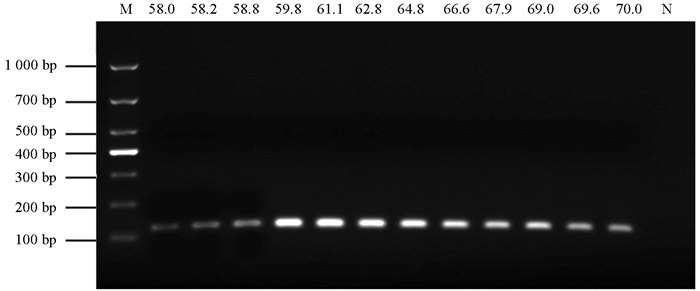
设计优化退火温度等条件,并应用Alphalmaher TM 2200软件对扩增结果进行分析,结果表明,在58~70 ℃的退火温度下,引物21F/21R对生姜青枯菌DNA模板均可扩增出单一条带(图 5). 当退火温度为61.1 ℃时,相对PCR效率最高(图 6). 结合不同延伸时间、循环次数的PCR扩增检测条件的试验结果,定PCR最优反应条件(总体积为25 μL)为:2×Taq Master PCR Mix 12.5 μL,引物21F/21R 0.5 μL,模板1 μL,补加dd H2O至25 μL. 反应程序为:94 ℃预变性5 min;94 ℃变性30 s,61.1 ℃退火30 s,72 ℃延伸30 s,30个循环;72 ℃延伸10 min.
-
特异性检测:按照优化后的反应条件,用引物21F/21R对不同种属来源的菌株基因组DNA模板进行PCR扩增,仅生姜青枯菌基因组DNA能扩增出1条125 bp的特异性条带,而其他菌株及阴性对照均无扩增条带(图 7). 通过对扩增产物进行测序,并经GenBank数据库中BLAST比对,扩增出的特异性片段与青枯雷尔氏菌的同源性高达99%,因此,引物21F/21R能够特异性地检测青枯雷尔氏菌菌株.
灵敏度检测:利用优化后的PCR反应条件对8个不同浓度梯度的模板DNA进行扩增,扩增结果如图 8所示. 随模板DNA浓度的降低条带亮度也在下降,引物21F/21R对供试菌株基因组的最低检测浓度为10-2 ng/μL.
-
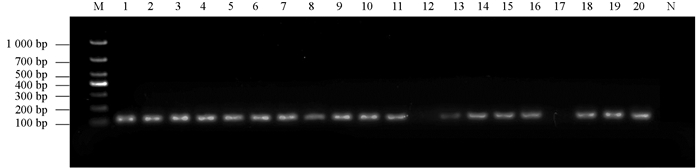
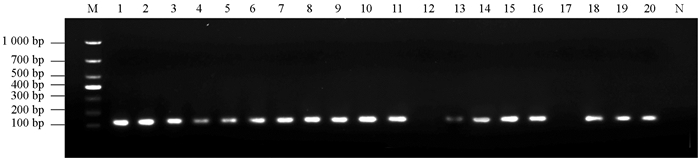
通过生姜青枯病特异性引物21F/21R对不同地区的病姜组织和土壤进行检测. 结果显示,20份从不同地区采集的病株组织中有2份病样组织未检测到目标菌(图 9).
土壤样本中,20份从不同地区采集的病株根际土壤中有2份病样组织未检测到目标菌,病菌检出率为90%(图 10). 此外,通过常规方法对病样组织和带菌土壤进行了病原菌分离培养和鉴定,其鉴定结果与PCR分子检测结果相吻合,由此可见,此方法可以检测感病生姜植株和土壤中的病原菌.
2.1. 生姜青枯病病原菌的分离
2.2. 致病性测定
2.3. 形态学鉴定
2.4. 分子生物学鉴定
2.5. 生理小种和生物型鉴定
2.6. PCR检测方法的建立及优化
2.7. PCR反应特异性及灵敏度检测
2.8. 发病植株和土壤的青枯病菌检测
-
生姜因长期采用无性繁殖的方式重茬种植,导致多种土壤病原菌不断积累,包括生姜青枯病在内的重大土传病害普遍发生,严重威胁广大姜农的经济收入[26]. 尽管自20世纪60年代开始,陈莉等[9]、戢俊臣等[10]和赵志祥等[11]对不同地区生姜青枯病病原菌进行了研究,但是有关湖北省内的生姜青枯病病原菌的研究却鲜有报道. 本研究从湖北省恩施土家族苗族自治州的生姜青枯病病株中分离得到了菌株ES202023,经致病性测定、形态学特征和分子生物学的鉴定,证明湖北恩施生姜青枯病的病原菌为青枯雷尔氏菌(R. solanacearum),与前人研究结果一致.
随着病原菌检测技术的不断发展,PCR分子检测技术已经较为成熟,在植物病原菌的检疫和田间预警中得到广泛的应用,为植物病害的有效防控提供了技术支持. 在植物病原菌的快速分子检测上,李华伟等[27]根据甘薯薯瘟病菌的特异基因(orf428)序列设计了重组酶聚合酶等扩增(RPA)引物,建立了能够快速检测甘薯薯瘟病菌的RPA方法. 李得铭等[28]对番茄青枯菌进行分离,并通过筛选3对特异性引物,建立三重PCR,检测番茄植株和土壤中的青枯菌. 张丽芳等[29]以烟草青枯菌和其他2个烟草病原菌的基因组DNA为模板,分别设计3对特异性引物,建立了能同时检测3种病害的多重PCR方法. 以上研究表明,利用单一或多重的PCR技术方法能有效检测病原菌的存在. 本研究构建的生姜青枯菌PCR快速分子检测技术,其PCR快速检测技术方法特异性强,能排除番茄青枯菌、土豆青枯菌、藿香青枯菌等不同生理小种的干扰. 通过优化检测引物PCR反应条件,灵敏度达到10-2 ng/μL,可以满足生姜生产中青枯菌检测的需求. 该方法不仅能检测植株的带菌情况,还可用于田间土壤青枯菌的检测,有助于生姜青枯病田间早期预警检测,为生姜青枯病的绿色防控提供科学依据.








 DownLoad:
DownLoad: