-
青蒿素是从青蒿(Artemisia annua L.)中分离的一种含有过氧桥基团结构的新型倍半萜内酯[1], 对于治疗脑型疟疾和抗氯喹恶性疟疾有特效[2].青蒿素联合治疗法(Artemisinin Combination Therapies, ATCs)是世界卫生组织(WHO)唯一推荐使用的基于青蒿素治疗疟疾的最有效的治疗方法[2].青蒿中青蒿素含量很低, 从天然青蒿中提取青蒿素远不能满足巨大市场需求[3], 提高青蒿素产量成为一项迫切任务.目前, 通过代谢工程提高青蒿素产量主要在两方面开展研究:采用合成生物学在酵母中合成青蒿酸并进而光氧化形成青蒿素[4]; 采用代谢工程技术培育青蒿素超高产青蒿新品种[5]. Keasling团队[6]在酵母中成功的实现了青蒿素的部分合成, 但是其现有的年产量有限且成本较高, 无法满足巨大市场需求.培育青蒿素超高产青蒿新品种仍然是很有前景的研究方向.开展青蒿素合成途径分子生物学研究是实现青蒿代谢工程育种的前体.甲瓦龙酸途径(MVA途径)为青蒿素生物合成提供基本5碳前体, 3-羟基-3-甲基戊二酰辅酶A还原酶(3-hydroxy-3-methylglutaryl-CoA reductase, HMGR)催化3-羟基-3甲基戊二酰辅酶A形成甲羟戊酸.在植物中, HMGR基因以家族形式存在, 提高HMGR表达能促进萜类积累[7].在青蒿中超量表达外源HMGR基因能显著提高青蒿素含量[8].然而, 关于青蒿HMGR家族研究未见报道, 各家族成员在青蒿素生物合成中的具体贡献也不清楚.
本研究以青蒿为研究对象, 根据我们和上海交通大学联合测定的青蒿全基因组序列(未公开), 结合RACE技术克隆青蒿HMGR家族基因, 并对其进行了生物信息学分析.通过实时荧光定量PCR技术, 对3条HMGR基因进行了组织表达特异性分析, 以及MeJA和损伤诱导的表达模式分析.通过不同层面的实验数据以及分析, 解析青蒿HMGR家族成员不同功能.
HTML
-
材料为具有相同遗传背景青蒿快繁幼苗(Xiang et al)[10].参照Wang等[12]和Liu等[13]的方法进行MeJA和损伤处理.将实验材料用液氮研磨, 提取RNA后反转录合成cDNA用于后续实验, 提取RNA和反转录方法参考Liu[13]等的方法.
-
根据青蒿全基因组、trichOME Database、NCBI上已经报道的青蒿HMGR基因片段, 利用Vector NTI suite 11.5软件进行拼接与比对找到3条不同的青蒿HMGR序列, 根据这3条序列分别设计3'RACE巢式PCR引物(表 1), 利用引物、dNTP和HiFi酶等按巢式PCR程序扩增获得AaHMGRs的3'RACE, 利用拼接结果设计5'RACE巢式PCR引物(表 1), 按照同样程序获得AaHMGRs的5'RACE, 并拼接获得AaHMGRs的电子全长, 用PCR扩增获得AaHMGRs的物理全长.
-
通过Vector NTI suite 11.5软件进行CDS的查找和翻译, 在NCBI网站的BLAST进行核酸和氨基酸序列比对; 用ClustalX软件进行氨基酸序列多重比对, 用MEGA4.1的Neighbor-joining进行进化树的构建.蛋白质的基本性质在ExPASy网站在线分析.
-
根据获得的AaHMGRs利用Beacon designer软件设计荧光定量特异性引物(表 2), EF1α为内参基因, 用染料iTaqTM Universal SYBR® Green Supermix通过iQ5 cycler荧光定量PCR仪反应并根据2-ΔΔCt方法计算得到AaHMGRs的相对表达量, 反应的条件为:预熔解95 ℃ 2 min, 熔解95 ℃ 15 s, 退火58.2 ℃ 20 s, 72 ℃延伸20 s, 循环40次, 熔解曲线, 55~95 ℃, 80个循环, 每循环升温0.5 ℃(Liu et al).
1.1. 植物材料以及处理
1.2. 基因克隆
1.3. 生物信息学分析
1.4. 表达分析
-
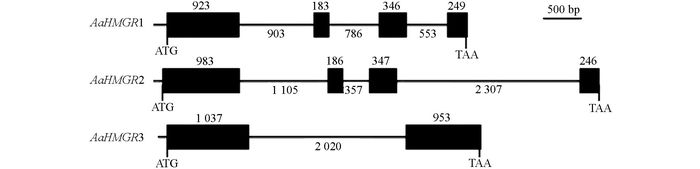
本研究克隆获得3条青蒿HMGR基因全长cDNA, 结果显示:AaHMGR1全长2 053 bp, 包含268 bp的3'UTR, 81 bp的5'UTR和1 704 bp的CDS, 编码1个568个氨基酸残基的多肽; AaHMGR2全长2 285 bp, 包含467 bp的3'UTR, 63 bp的5'UTR和1 755 bp的CDS, 编码585个氨基酸残基的多肽; AaHMGR3全长2 116 bp, 包含247 bp的3'UTR, 132 bp的5'UTR和1 737 bp的CDS, 编码579个氨基酸残基的多肽. AaHMGR1和AaHMGR2的cDNA相似度为62.4%, AaHMGR1和AaHMGR3的cDNA相似度为61.1%, 而AaHMGR2和AaHMGR3的cDNA相似度为66.1%, 说明这3条AaHMGR是不同的序列.再将其cDNA与青蒿基因组对比后发现这3条AaHMGR均能够找到对应的基因组序列, 它们编码区的基因组结构不同(图 1). AaHMGR1和AaHMGR2结构相似, 都具有4个外显子和3个内含子, 但是外显子长度略有差异. AaHMGR3只有1个外显和2个内含子.这3条AaHMGR在cDNA的3'UTR, 5'UTR、编码区和基因组序列上均有较大差异, 表明是3条不同的HMGR基因.
-
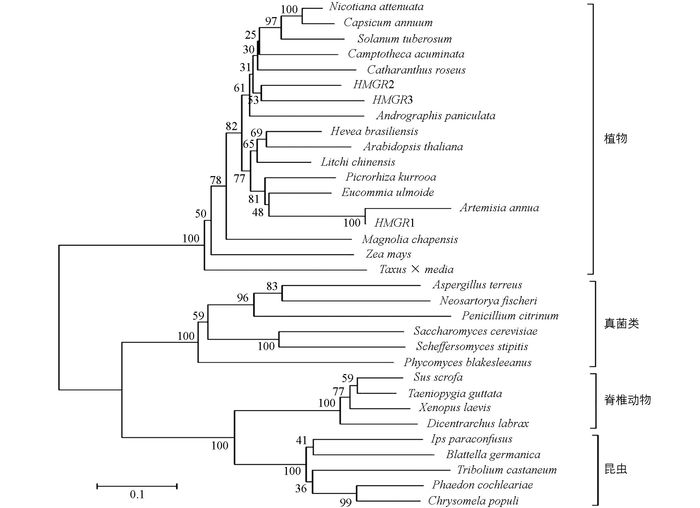
BLASTP分析结果表明这3条AaHMGR报道的HMGR具有较高相似性, 属于HMGR类蛋白质.这3条AaHMGR在氨基酸序列上高度相似, 且具有十分接近的分子量和等电点(表 3).在3条AaHMGR中, 均能够找到2个HMG-CoA结合基序和两个NADP(H)结合基序.氨基酸序列多重比对结果(图 2)显示:本研究克隆获得的AaHMGRs与其他物种的HMGR具有较高同源性, AaHMGR1与杜仲(AAV54051)、胡黄连(ABC74565) 的相似性分别为73.3%, 75%, AaHMGR2与长春花(AAT52222) 的相似性为71.3%, AaHMGR3与穿心莲(AAL28015) 的相似性为68.1%, 该结果与之前的研究结果相一致[9].从氨基酸的多重比对结果可以确认本研究所克隆的3条序列均为HMGR.利用MEGA4.0 Neighbor-joining方法构建进化树.结果显示:在进化关系上, AaHMGRs属于植物HMGR家族, AaHMGR1单独被分在一支, 与已报道的青蒿的HMGR(AAD47596) 属于同一条[8], 这个结果与氨基酸的多重比对结果一致, 而AaHMGR2, AaHMGR3则位于进化树的同一分支上, 与AaHMGR1的亲缘关系较远(图 3).
-
虽然基因家族不同成员编码同一类蛋白质而具有相同的蛋白质功能, 但是家族成员可以通过基因表达的不同时空特异性进行分工.我们采用qPCR分析了这3个AaHMGR组织表达谱、对MeJA和机械损伤的响应, 从而初步探讨它们的不同分工.
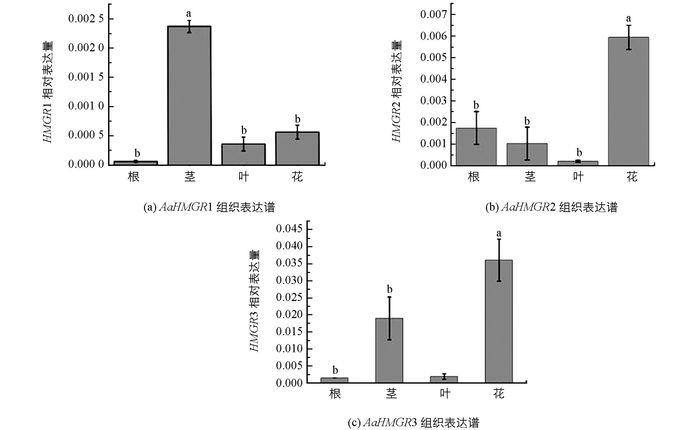
分析3个HMGR在青蒿的组织中相对表达量, 结果显示:AaHMGR1只在青蒿地上部分表达, 在根中不表达, 在茎中表达量高于叶和花中表达量.而AaHMGR2和AaHMGR3在花中相对表达量最高(图 4).结果表明, AaHMGR1, AaHMGR2, AaHMGR3在植物组织中表达模式有差异, 推测3条HMGR在青蒿中的功能不一样.
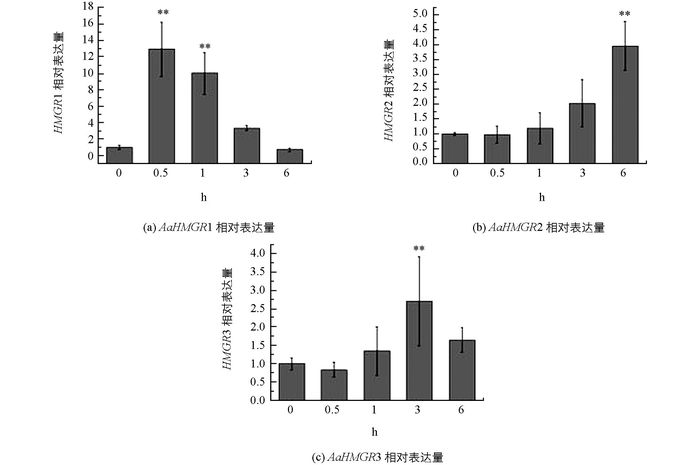
分析MeJA处理后青蒿的3个HMGR的表达水平, 结果表明:3个AaHMGR对MeJA处理均有响应, 但其响应模式存在较大的差异. AaHMGR1对MeJA处理响应比较迅速, 处理后0.5 h其表达量就达到最大值, 其后逐渐下降.而且0.5 h和1 h时基因表达量上调, 且对照相比均呈现差异有统计学意义(p < 0.01), 是对照的12.95倍和10.02倍.这个结果与已报道的研究结果相符[10].用MeJA处理青蒿并检测HMGR(AF142473) 的表达量及青蒿素的含量, 结果显示HMGR的表达量在处理1 h后显著提高, 并且青蒿素的含量在1 h后也相应提高, 暗示HMGR可能参与青蒿素的合成. AaHMGR2在MeJA处理后, 相对表达量整体呈现逐渐上升的趋势, 但是其响应的时间则没有AaHMGR1迅速, 6 h时其表达量的上调幅度与对照相比差异达到统计学意义(p < 0.01). AaHMGR3在MeJA处理后, 相对表达量整体轻微响应, 3h时达到最大值, 是对照的2.71倍(p < 0.01) (图 5).结果表明, AaHMGR1, AaHMGR2, AaHMGR3均受MeJA的诱导, 但AaHMGR1的响应更强烈迅速, 因此推测AaHMGR1可能直接参与青蒿素的合成.
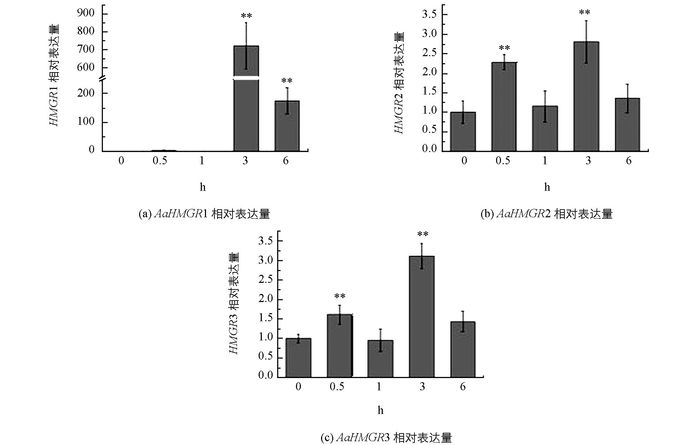
青蒿的HMGR家族基因在损伤处理后的基因表达情况显示:青蒿的3条HMGR均对损伤处理有不同程度的响应. AaHMGR1在损伤处理后, 在3h时达到最大值, 是对照的724倍(p < 0.01), 其后在6 h时表达量有所下降, 这个结果与先前的研究Liu等[13]结果相似.而AaHMGR2和AaHMGR3基因在损伤处理后表达模式基本一致, 呈现波动式的变化.仅从最大表达量来看3个基因具有一致性, 都是在3h达到最大值, 但是上调幅度却不同, AaHMGR1上调幅度较大.由此可知, AaHMGR1, AaHMGR2, AaHMGR3均受损伤诱导, 但是AaHMGR1的响应更强烈(图 6).因此推测AaHMGR1可能在青蒿素的生物合成途径中起着更为重要的作用.
2.1. AaHMGRs全长cDNA克隆
2.2. 青蒿HMGR生物信息学分析
2.3. AaHMGRs表达分析
-
本研究获得的AaHMGR1, AaHMGR2, AaHMGR3与其它已经报道的植物HMGR具有高度同源性, 且均包含有HMGR所特有的HMG-CoA和NADP(H)保守结构域[9]. 3条HMGR的DNA结构不相同, 且在组织中表达中AaHMGR1在茎中表达最高, 而AaHMGR2和AaHMGR3在花中表达最高, 说明AaHMGR2和AaHMGR3的本底水平表达比AaHMGR1高, 更近一步说明可能在激素或损伤诱导时AaHMGR1更有利于青蒿素生物合成.实验结果显示在MeJA和损伤处理后AaHMGRs基因的响应模式确有统计学差异, AaHMGRs基因受MeJA和损伤诱导, 特别是受损伤诱导, 与先前的研究结果一致:Liu等[13]用损伤处理青蒿后检测HMGR(AF142473) 的表达量及青蒿素含量, 在处理后HMGR基因表达量在2h达到最大值, 青蒿素含量在2h后显著提高并在4h时达到最大值.可利用损伤青蒿作为提高青蒿素的有效方法之一. MeJA在短时间处理后, AaHMGR1的表达量显著提高, 结果与Xiang[10]的研究结果相一致, 可考虑尝试利用AaHMGR1来提高青蒿素的生物合成.青蒿素的含量在MeJA处理后也得到显著提高, 这可能是MeJA的处理, 激活了参与信号转导同时参与青蒿素合成的转录因子的表达, 从而进一步引起下游参与青蒿素合成的基因的表达, 最终导致青蒿素含量的提高.目前研究发现很多转录因子均参与MeJA的信号转导, 并启动下游基因的表达[11].损伤处理后, AaHMGR1的表达量和青蒿素的含量显著提高, 这可能是处理后, 诱导了参与损伤信号转导同时也参与青蒿素合成的转录因子表达, 从而进一步引起下游参与青蒿素合成的基因的表达, 最终导致青蒿素含量的提高.但是关于损伤所涉及的转录因子的研究还较少, 因此这方面的研究还需要再深入, 探寻其提高青蒿素含量的分子机制.






 DownLoad:
DownLoad: