-
开放科学(资源服务)标志码(OSID):

-
海藻糖是一种非还原性双糖,广泛存在于细菌、真菌、昆虫和植物等许多生物体内[1]. 海藻糖酶能将一分子海藻糖水解为两分子葡萄糖,通过糖酵解为各个组织器官提供能量,或为几丁质合成提供原料,因而海藻糖酶在昆虫体内起着非常重要的作用[2]. 根据是否含有跨膜结构域将昆虫海藻糖酶基因分为两类:①可溶型海藻糖酶(Tre1),主要分解细胞内的海藻糖;②膜结合型海藻糖酶(Tre2),主要水解食物中的海藻糖[3-4]. 海藻糖酶是海藻糖分解代谢的关键酶,广泛参与并调控昆虫的生长发育、非生物胁迫以及激素反应等,与昆虫能量代谢、几丁质合成和诱导滞育密切相关. 目前,已经在甜菜夜蛾Spodoptera exigua[5]、赤拟谷盗Tribolium castaneum[6]、飞蝗Locusta migratoria[7]和灰飞虱Laodelphax striatellus[8]等昆虫中,通过RNAi技术证实了海藻糖酶基因在能量代谢和几丁质合成方面发挥着重要作用. Chen等[5]通过显微注射法对甜菜夜蛾海藻糖酶基因SeTre1和SeTre2进行RNAi研究发现,基因转录水平及几丁质含量均显著降低,且SeTre1和SeTre2分别在昆虫的表皮和中肠的几丁质合成中起重要作用. 张倩等[8]通过饲喂法干扰灰飞虱海藻糖酶基因LSTre1和LSTre2,发现基因转录水平分别降低49%和41%,并导致昆虫体质量减轻、死亡率显著升高. 由于哺乳动物没有海藻糖代谢系统,以海藻糖酶为靶标的新农药对人畜等非靶标生物可能不存在毒害作用[8-9],因此海藻糖酶便成为新型高效杀虫剂设计和开发的潜在优良靶标.
豌豆蚜Acyrthosiphon pisum是影响粮食生产的重要害虫之一,主要通过取食植物汁液、诱发煤污病和传播植物病毒的方式[10-12]为害农作物. 目前,关于豌豆蚜海藻糖酶的RNA干扰研究尚未见相关报道. 本研究以豌豆蚜为试虫,通过基因克隆得到可溶型海藻糖酶基因ApTre-1的全长编码序列,运用生物信息学方法对ApTre-1基因和蛋白氨基酸序列进行序列特征分析,构建系统进化树分析豌豆蚜海藻糖酶ApTre-1的进化关系,利用荧光定量PCR(qPCR)技术明确ApTre-1基因在豌豆蚜不同生长发育阶段的表达水平;在上述基础上,进一步通过显微注射法和饲喂法测定两种dsRNA片段对海藻糖酶ApTre-1基因的干扰效率,以及对试虫生长发育的影响和致死效应. 本研究对基于RNAi技术的海藻糖酶基因在害虫防治中的应用进行了初步探索,可为今后深入研究提供有价值的前期参考.
HTML
-
豌豆蚜由西北农林科技大学应用昆虫学重点实验室提供,饲养于人工气候培养箱内的盆栽蚕豆(“晋农”精选蚕豆)上,饲养条件:温度(21±1) ℃,相对湿度70%~75%,光照周期L(光照)∶D(黑暗)=16∶8.
-
按照TRNzol Universal Reagent试剂盒(天根)说明书,对豌豆蚜3龄若虫进行总RNA提取. 取1 μg总RNA作为模板,按照HiFiScript gDNA Removal cDNA Synthesis Kit试剂盒(康为世纪)说明书,去除基因组DNA后,反转录合成cDNA第一条链.
基于NCBI(National Center for Biotechnology Information)数据库中豌豆蚜的基因组序列,本研究利用大豆蚜Aphis glycines海藻糖酶基因(GeneBank登录号:JQ246351.1)进行BLAST搜索,获得豌豆蚜全基因组数据中编号为Contig13577的序列(GeneBank登录号:ABLF02013269.1). 通过软件进一步分析找到豌豆蚜海藻糖酶编码基因阅读框后,利用Primer Premier 5软件设计特异性引物(表 1),以豌豆蚜cDAN第一链为模板,扩增豌豆蚜的海藻糖酶编码基因. PCR反应条件:98 ℃预变性1 min,98 ℃变性10 s、55 ℃退火30 s、72 ℃延伸3 min,共35个循环. 扩增产物经1%琼脂糖凝胶检测后,进行回收纯化. 纯化后的DNA通过DNA A-Tailing Kit(TaKaRa)加“A”反应后,与pMD 19-T Vector(TaKaRa)连接,构建重组质粒. 将重组质粒转入大肠杆菌感受态细胞Escherichia coli DH5α,菌落PCR鉴定正确后,送至生工生物工程(上海)股份有限公司(Sangon Biotech)测序.
-
利用在线软件Softberry(http://www.softberry.com)分析豌豆蚜基因组中海藻糖酶基因的内含子和外显子组成;利用在线软件ExPASy(https://web.expasy.org/protparam)预测蛋白质的相对分子质量和理论等电点;通过在线软件Signal P(https://services.healthtech.dtu.dk/)和TMHMM(https://services.healthtech.dtu.dk/)分别对蛋白质的信号肽和跨膜区域进行分析;通过NCBI在线工具BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行蛋白质同源搜索,应用MEGA 6软件中的Clustal W进行多重序列比对,采用邻位相连法(Neighbor-joining)进行聚类分析,构建系统进化树. 重复次数为1 000次,树枝上的数字表示bootstrap验证中该树枝可信度百分比大于50%的数值(图 1).
-
采用qPCR技术分析ApTre-1基因在豌豆蚜不同龄期(1龄、2龄、3龄、4龄若蚜和无翅成蚜)的表达水平. 根据获得的豌豆蚜ApTre基因序列设计qRCP特异性引物(表 1),选择EF-1a和RPS-20作为双内参基因[13]. 以豌豆蚜1龄若虫的相对表达量为基准,以无核酸酶水代替cDNA为阴性对照,以不加反转录酶的核酸代替cDNA排除核酸样品中基因组DNA污染的可能性. qPCR反应体系(20 μL)为:5倍稀释的cDNA模板2 μL,上、下游引物各0.8 μL(10 mmol/L),2×TB Green Premix Ex Taq Ⅱ 10 μL,无核酸酶水6.4 μL. 反应于Roche Light Cycler 480实时荧光定量PCR仪(德国Roche Diagnostics GmbH)上进行. qPCR反应条件为:95 ℃ 5 min,95 ℃ 5 s,60 ℃ 1 min,共40个循环. 根据熔解曲线,确定引物及扩增特异性. 实验设置3个生物学重复,每个生物学重复设置3个技术重复. 采用2-ΔΔCt法计算基因的相对表达量[14].
-
以序列验证无误的ApTre为模板进行PCR扩增,引物见表 1. 使用Gel Extraction Kit试剂盒(天根)对PCR产物进行纯化回收. 根据T7 RiboMAXTM Express RNAi System试剂盒(Promega)说明书,进行体外dsRNA合成. 取2 μL dsRNA稀释10倍后,使用分光光度计测定dsRNA的浓度,并通过1%的琼脂糖凝胶进行检测,置于-80 ℃冰箱保存.
-
参照叶超[15]的方法,选择生长健康、状态一致的4龄若蚜,使用显微注射器分别注射2种不同的dsApTre-1片段,以dsGFP为对照. 每只蚜虫注射dsRNA(1 μg/μL)60 nL,每个处理10只蚜虫,设6个生物学重复. 注射完成后,将豌豆蚜放入培养皿中新鲜的蚕豆叶片上饲养. 采用qPCR技术检测注射试虫2 d后的ApTre-1基因相对表达水平.
-
根据Auclair等[16]蚜虫人工饲料配方配置豌豆蚜的人工饲料,采用饲蚜器进行饲喂. 饲蚜器为两端开口的玻璃管,将封口膜(美国Parafilm)拉至最薄,附于玻璃管的一端,使用移液枪将40 μL的人工饲料滴加至封口膜中央(dsRNA的浓度为1 μg/μL),然后再拉伸一张封口膜盖在人工饲料上,使人工饲料均匀平铺于两膜之间. 将饲蚜器置于智能人工光照气候培养箱中,每2 d更换1次人工饲料. 每个处理10头若蚜,重复6次. 每天观察并统计存活数,每2 d称质量记录1次豌豆蚜体质量. 采用qPCR技术检测饲喂2 d后ApTre-1基因的相对表达水平.
1.1. 供试昆虫
1.2. 豌豆蚜海藻糖酶基因的克隆
1.3. 豌豆蚜海藻糖酶蛋白ApTre-1生物信息学分析
1.4. 实时荧光定量PCR检测ApTre-1基因在不同龄期的表达水平
1.5. 体外合成dsRNA
1.6. 显微注射法测定ApTre-1基因不同dsRNA片段的RNAi效率
1.7. 饲喂法测定ApTre-1基因dsRNA片段的RNAi效应检测
-
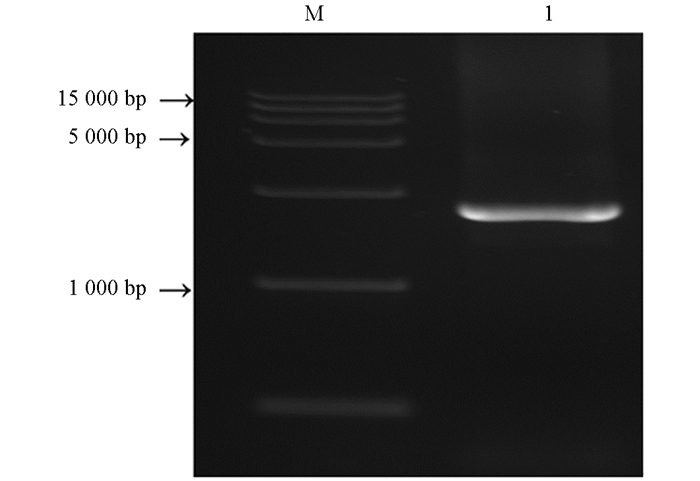
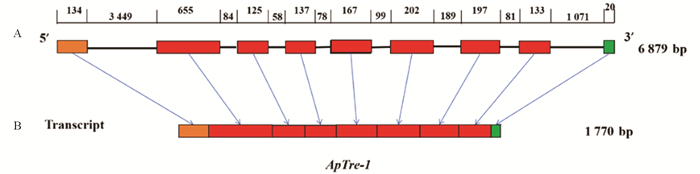
利用特异性引物对豌豆蚜海藻糖酶基因进行PCR扩增,扩增产物经1%琼脂糖凝胶电泳检测,获得1条大小约为2 300 bp的电泳条带(图 2). 将目的条带回收纯化后,经连接、转化和测序,筛选阳性克隆,得到海藻糖酶基因(ApTre-1)的cDNA序列. 通过Vector NT软件对PCR扩增序列进行分析,确定了豌豆蚜海藻糖酶基因的开放阅读框. 该基因的开放阅读框(ORF)为1 770 bp,编码589个氨基酸. 在基因组中DNA水平上该基因由9个外显子和8个内含子组成(图 3). 其编码的氨基酸序列与NCBI中注释和预测的豌豆蚜海藻糖酶氨基酸序列(GenBank登录号:XM001950229.4)完全一致.
使用在线软件ExPASy预测豌豆蚜海藻糖酶ApTre-1蛋白的相对分子质量为6.83×104;理论等电点(PI)为6.10. 利用Signal P在线预测该蛋白的信号肽,结果表明ApTre-1蛋白的N段含有一段长为20 aa的信号肽序列. 利用TMHMM预测该蛋白的跨膜结构域,结果表明ApTre-1蛋白同其他已知昆虫的可溶型海藻糖酶一样无跨膜区域. 通过NetNGlyc软件预测,发现ApTre蛋白含有4个糖基化位点,分别位于124,234,346和473位氨基酸. 通过GeneDoc软件进行蛋白质同源序列比对,发现ApTre-1蛋白含有“PGGRFRELYYWDTY” “QWDFPNAWPP”2个标签序列和一个富甘氨酸区域“GGGGEY”. 并且,发现在蚜虫等半翅目昆虫中还含有“YYLNRSQPP” “PRPESYREDY” “IIPVDLN” “MFEKYD”和“GFGWXNG”共5个高度保守区域(图 4).
-
通过ClustalW软件将豌豆蚜ApTre-1蛋白与其他已知的13种昆虫海藻糖酶蛋白氨基酸序列进行同源比对,利用MEGA 6软件中的Neighbor-Joining方法进行分子系统进化分析,得到昆虫海藻糖酶蛋白分子进化树(图 1). 从该系统发育树可以看出,海藻糖酶分为可溶型海藻糖酶和膜结合型海藻糖酶两大类,在分类上属于同一目的昆虫均单独形成一个亚分支,这与传统的分类结果一致. 本实验克隆得到的豌豆蚜ApTre-1蛋白与大豆蚜、飞虱等可溶型海藻糖酶归为一个亚分支,其中豌豆蚜ApTre-1蛋白与大豆蚜Aphis glycines的可溶型海藻糖酶(AgTre-1)聚在更小的一个分支内,表明其氨基酸序列相似性较高,与烟粉虱Bemisia tabaci、褐飞虱Nilaparvata lugens及白背飞虱Sogatella furcifera可溶型海藻糖酶的氨基酸序列相似性则相对较低.
-
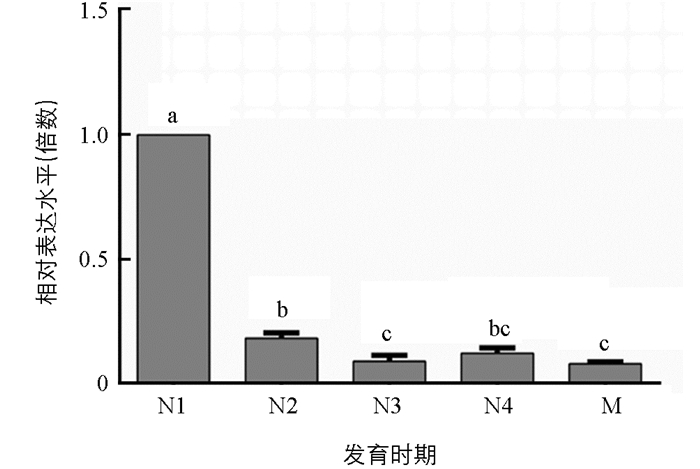
通过qPCR技术,对ApTre-1基因在豌豆蚜1~4龄若虫和成虫中的表达水平进行检测分析(图 5). 结果表明:ApTre-1基因在豌豆蚜整个生长发育期间均有表达,但不同发育阶段表达量具有明显差异,其mRNA在1龄若蚜中表达水平最高,显著高于其他各阶段,约为2龄若蚜的5倍、成虫的12倍.
-
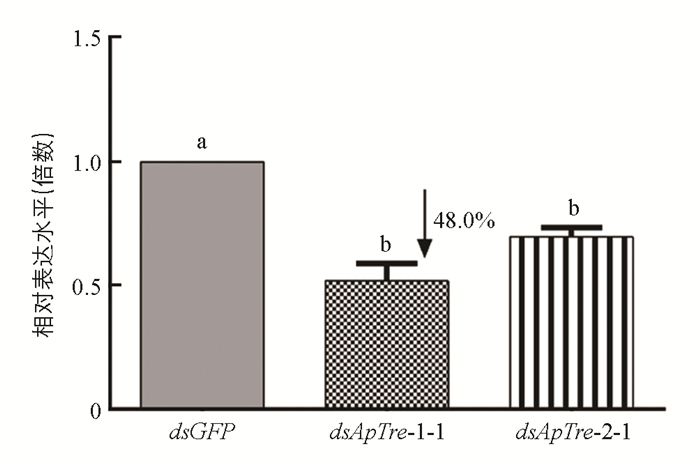
对4龄若蚜进行显微注射dsRNA后,放入培养皿中用新鲜蚕豆叶片进行饲喂,2 d后采用qPCR技术检测蚜虫体内ApTre-1基因表达量的变化(图 6),以绿色荧光蛋白(GFP)的dsGFP为对照. 由图 6可见,经过显微注射两种dsRNA后,蚜虫体内ApTre-1基因转录水平均受到不同程度的抑制. 在分别注射dsApTre-1-1和dsApTre-1-2片段2 d后,ApTre-1基因的相对表达量分别降低了48.0%和30.3%.
-
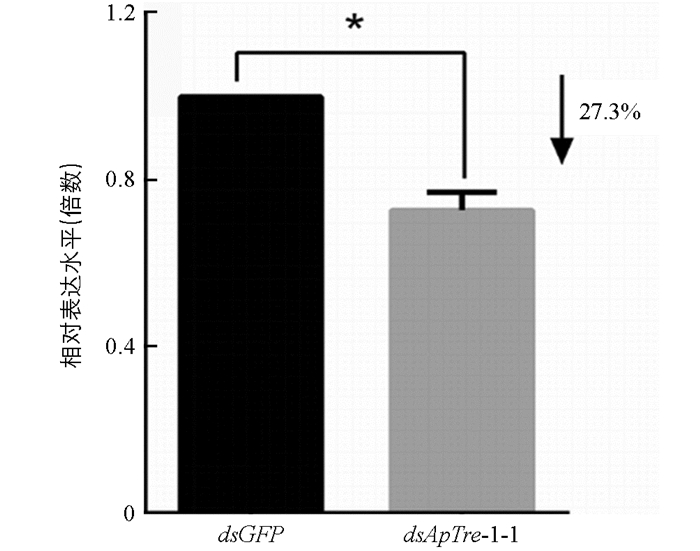
根据以上结果,选择其中干扰效率较高的dsApTre-1-1片段与人工饲料混合,进一步采用饲喂法测定其对靶基因ApTre-1的RNA干扰效率. 以取食含dsGFP饲料的蚜虫为对照,检测取食含dsApTre-1-1饲料2 d后的蚜虫体内可溶型海藻糖酶基因表达量的变化(图 7). 从图 7中可以看出,蚜虫取食含dsApTre-1-1的饲料后与对照组(含dsGFP饲料)试虫相比,ApTre-1基因表达量降低了27.3%,差异具有统计学意义(p<0.5%).
-
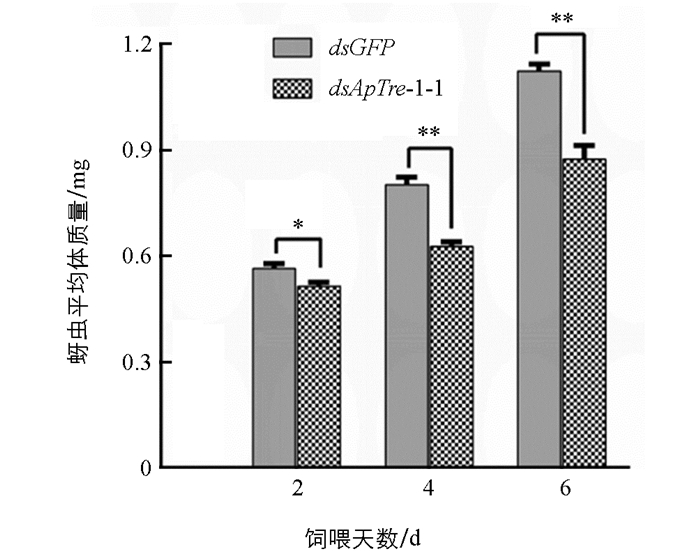
以饲喂含有质量浓度为1 μg/μL dsGFP和dsApTre-1-1的饲料分别作为非靶标对照组和处理组,统计第0,2,4,6 d存活豌豆蚜的平均体质量(图 8). 从图 8中可以看出,连续饲喂含有dsApTre-1的饲料2 d后,试虫平均体质量与对照组相比开始显著降低. 到第6 d时,蚜虫实验组平均体质量为0.88 mg,与对照组的平均体质量(1.13 mg)差异具有统计学意义(p<1%),表明试虫取食dsApTre-1-1片段后,生长发育受到明显抑制.
-
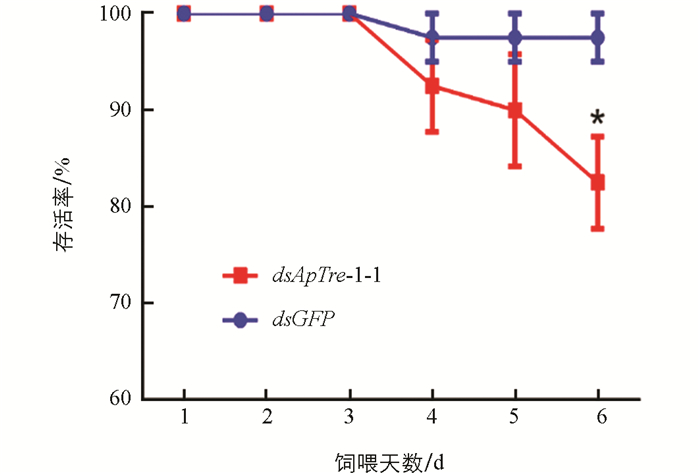
以饲喂含有质量浓度为1 μg/μL dsGFP和dsApTre-1-1的饲料分别作为非靶标对照组和处理组,统计2龄若蚜取食dsRNA后1~6 d的存活率(图 9). 由图 9可以看出,连续饲喂含有dsApTre-1-1饲料3 d后,蚜虫的存活率开始降低,到第6 d时蚜虫的存活率为82.5%,与对照组之间差异具有统计学意义(p<0.05).
2.1. 豌豆蚜海藻糖酶基因ApTre-1克隆及序列分析
2.2. 豌豆蚜ApTre-1蛋白的分子进化分析
2.3. 豌豆蚜海藻糖酶基因ApTre-1在不同发育时期的表达分析
2.4. 豌豆蚜海藻糖酶基因ApTre-1的RNA干扰效率
2.5. 饲喂dsApTre-1-1片段对豌豆蚜RNA干扰效率及生长的影响
2.5.1. 饲喂dsApTre-1-1片段对豌豆蚜ApTre-1基因的RNA干扰效率
2.5.2. 饲喂dsApTre-1-1片段对豌豆蚜生长的影响
2.5.3. 饲喂dsApTre-1-1片段对豌豆蚜的致死率
-
海藻糖酶是昆虫体内重要的调控酶,自从1992年和2005年分别鉴定出黄粉虫Tenebrio molitor的可溶型海藻糖酶[17]和家蚕的膜结合型海藻糖酶[3]后,学者们很快就发现昆虫普遍具有这两种类型的海藻糖酶. 本研究基于已公布的豌豆蚜基因组数据库,扩增获得了豌豆蚜可溶型海藻糖酶基因ApTre-1,其全长为1 770 bp,编码589个氨基酸,与NCBI中预测的豌豆蚜海藻糖酶基因(GenBank登录号:XM001950229.4)氨基酸序列完全一致,揭示了海藻糖酶氨基酸序列在不同地域豌豆蚜中的高度保守性. 序列分析表明ApTre-1氨基酸序列具有典型的标签序列和富甘氨酸序列,系统进化树揭示其与大豆蚜海藻糖酶基因序列相似性最高,并且与其他半翅目昆虫,包括褐飞虱、白背飞虱以及烟粉虱的可溶型海藻糖酶聚为一类.
海藻糖酶基因在豌豆蚜体内的表达水平处于动态变化过程. Bansal等[18]研究发现,海藻糖酶基因在大豆蚜1,2龄若蚜期的表达水平较高,随后其表达水平持续降低,在成蚜中表达水平最低. 本研究中,可溶型海藻糖酶基因ApTre-1在豌豆蚜不同龄期表达水平差异具有统计学意义,其在1龄若蚜期的表达量最高,且显著高于其他各生长发育阶段. 与文献数据相比,本研究中ApTre-1基因在豌豆蚜不同龄期的表达模式与其在大豆蚜不同龄期的表达模式较为一致,均在1,2龄若蚜期表达水平最高. 此外,在飞蝗研究中发现,海藻糖酶基因LmTre-1在卵发育前期和中期表达量极低,在末期表达量显著增加,在卵期最后1天表达量最高且显著高于其他时期[7]. 结合本研究,推测ApTre-1基因在豌豆蚜1龄若虫中的高表达,可能是因为1龄若蚜刚开始取食且处于快速生长发育阶段,与需要大量的能量有关. 但是,该推测尚需进一步开展相关研究工作去证实.
RNAi的效率与试虫体内靶基因的选择密切相关,因此在利用RNAi技术防治害虫的研究中需要从害虫体内筛选和鉴定高效靶标基因[19-20]. 目前研究较多的靶基因主要包括致死基因、抗性和免疫基因、生长发育相关基因和产卵相关基因等[21]. 例如,Terenius等[22]通过对鳞翅目昆虫RNAi研究结果进行比较发现,免疫相关基因的干扰效率显著高于其他类型的基因. 此外,基于已发现的害虫体内的高效靶基因,学者们利用植物表达这些靶基因的dsRNA来防治害虫已经取得了较大进展. 例如,植物介导针对几丁质合成酶基因(CHS)的RNA干扰技术已经在麦长管蚜Sitobion avanae[23]中进行了较为深入的研究,发现试虫取食表达dsRNA的第3代转基因小麦后,其体内的靶基因CHS1表达水平下降了45%~50%,试虫总蜕皮率也显著下降. 在褐色橘蚜Toxoptera citricida[24]的研究中,也发现取食植物传导的dsRNA后,试虫体内CHS的表达水平下降了48%,且多数试虫不能蜕皮进入到下一个龄期. Mao等[25]研究发现,桃蚜Myzus persicae连续取食植物表达的间隙基因(Mphb)dsRNA后,降低了靶基因的表达量,抑制了桃蚜的繁殖.
针对靶基因不同区域设计的dsRNA片段也会导致干扰效率的显著差异性. 本研究首先采用显微注射法针对靶标基因ApTre-1不同区域进行RNAi效率分析,发现dsApTre-1-1,dsApTre-1-2对靶基因ApTre-1沉默效率分别为47.7%和30.3%. Chen等[5]针对甜菜夜蛾2种海藻糖酶的RNAi研究发现,dsSeTre-1和dsSeTre-2片段注射试虫24 h后,2种海藻糖酶靶基因的沉默效率从50%逐渐上升,至72 h时达到最高(近80%). 相较而言,本研究中针对豌豆蚜海藻糖酶基因ApTre-1的RNAi效率相对较低,主要原因可能是本研究中所设计的2种dsRNA片段长度或位置不是最佳所致,其他原因尚需进一步分析研究.
目前,实验室研究中dsRNA导入昆虫体内的方法主要为显微注射法和饲喂法. 由于显微注射法过程较为繁琐、处理样本量少,较适用于昆虫基因功能的研究;而饲喂法过程较为简单,更接近昆虫的自然取食过程,有助于最终在实践生产中应用. 因此,饲喂法的应用实践性显著优于显微注射法. 在采用显微注射法分析的基础上,本研究选用其中RNA干扰效率较高的片段dsApTre-1-1进一步通过饲喂法测定其对豌豆蚜的RNA干扰效应,结果发现dsApTre-1-1对靶基因ApTre-1的沉默效率较注射法有较明显下降,靶基因的表达水平下降了27.3%,试虫第6 d时死亡率为17.5%. 张倩等[8]采用饲喂法对灰飞虱的2种海藻糖酶基因(LSTre-1和LSTre-2)进行RNA干扰效应研究,发现dsTre-1和dsTre-2对靶基因的干扰效率分别为49.1%和41.5%,在第2 d时致死率为10%~15%,以后逐渐上升,到第5 d时试虫死亡率为20%~40%. 二者相比,采用饲喂法测定的dsRNA干扰效率及致死率略高于本实验结果,但总体上致死率也相对较低. 上述研究结果说明在不同种类的试虫中,与显微注射法相比,昆虫取食dsRNA后对靶基因的干扰效率及致死率均相对较低很可能是一种普遍现象. 已有诸多研究表明,昆虫肠道中含有丰富的核酸酶,能够降解dsRNA,从而影响RNAi效率,并对通过饲喂方式让dsRAN进入试虫体内后保持稳定和传递极为不利[26-27],这可能是导致上述研究中通过饲喂法测定dsRNA的RNAi效率降低和致死率较低的重要原因之一. 据文献报道,针对靶基因不同长度的dsRNA片段也可能导致不同的RNA干扰效应,包括靶基因沉默效率、试虫生长发育、死亡与繁殖率等. 因此,在本实验的后续研究中,仍需针对靶基因设计合成更多不同类型的dsRNA片段,以便能筛选出对靶基因干扰效率更高的dsRNA片段,为针对海藻糖酶的RNAi技术应用于害虫防治提供有价值的参考.
本研究获得了豌豆蚜可溶型海藻糖酶基因(ApTre-1)序列,明确了其基因结构特点及在豌豆蚜不同生长发育阶段的表达差异,探索了显微注射和饲喂ApTre-1基因的dsRNA片段对豌豆蚜RNA的干扰效应,为今后以ApTre-1基因为靶标的RNA干扰技术在害虫综合防治中的应用奠定了基础.







 DownLoad:
DownLoad: